Application Note SpectraMax MiniMax® 300イメージングサイトメーターと
SpectraMax i3マルチモード検出プラットフォームによるミトコンドリア毒性の評価
- StainFreeテクノロジーで核の対比染色をせずに細胞画像を解析
- SoftMax Proソフトウェアの画像解析機能により、細胞単位でミトコンドリア染色を測定
- 重要なミトコンドリア毒性結果を迅速に得る
PDF版(英語)
はじめに
薬物スクリーニング計画において重要なことは、ミトコンドリアの健康状態に対する候補薬剤の影響をモニターすることである。ミトコンドリア活性は、様々な色の蛍光色素を用いて容易に分析できる。これらはしばしばDAPIのような核カウンターステインと併用され、画像中のすべての細胞の同定を可能にする。イメージングシステムの1つの蛍光チャンネルで核染色によりセルが同定されると、ミトコンドリア活性は、使用された特異性ミトコンドリア色素に対応する2番目のチャンネルの蛍光として、細胞ごとに測定することができる。しかし、核の対比染色は、実験ワークフローに一連の面倒なステップを追加し、結果を遅らせる。Molecular Devices社のStainFree™ テクノロジーは、SpectraMax® MiniMax™ 300イメージングサイトメーターの透過光チャンネルでイメージングした細胞を識別することで、研究者を核染色の煩わしさから解放します。
このアプリケーションのハイライトでは、SpectraMax® i3 Multi-Mode Detection Platformを使用して、細胞イメージングでミトコンドリア毒性を評価する方法を紹介します。イオノフォア抗生物質であるバリノマイシンを用いてPC12細胞のミトコンドリア機能を破壊し、MitoTracker Deep Red FMを用いてその影響をモニタリングした。細胞を同定するためにStainFreeと蛍光核色素を用いた解析を比較し、SoftMax® Proソフトウェアを用いて結果を評価した。SpectraMax® MiniMax® 300イメージングサイトメーターとSoftMax Proソフトウェアは、毒性の可能性のある化合物に反応したミトコンドリア活性を迅速に定量するための画像取得と解析のための完全なソリューションを提供します。
細胞ベースアッセイのセットアップ
PC12ラット褐色細胞腫細胞を、2.5%ウシ胎児血清、15%ウマ血清、1%ペニシリン/ストレプトマイシンを添加したF-12K培地で培養した。黒壁透明底96ウェルプレートに、セル10,000個/ウェル、100μLずつ播種した。セルは一晩接着させ、増殖させた。
播種の翌日、バリノマイシンを1μMから1nMまで1:2希釈系列でマイクロプレート中の細胞に添加した。プレーティングは37℃で24時間行った。化合物のインキュベーション終了後、ウェル内の培養液と化合物を、培養液で希釈したMitoTracker Deep Red FM(MTDR)の100 nM溶液で置換した。この試薬は生細胞中の活性ミトコンドリアを標識し、MiniMax サイトメーターの赤色蛍光チャンネルで検出される遠赤色波長で蛍光を発する。
セルを30分間インキュベートした後、染色を除去した。その後、細胞を4%パラホルムアルデヒドで固定した。固定した細胞の核をEarlyTox Dead Green Dye(Molecular Devices社、商品番号R8216)でカウンター染色した。固定は StainFree 分析には必要ないが、比較のために本実験の全セルに対して行った。細胞はMiniMaxサイトメーターの透過光(TL)チャンネルでイメージングし、細胞数の測定と形態の評価を行い、赤色蛍光チャンネルではミトコンドリア活性を測定した。また、個々の細胞の核を同定し、StainFree細胞数と並行して比較解析を行うために、緑色チャンネルで細胞をイメージングした。
SoftMax® Proソフトウェアを用いて、2つの異なる画像解析をセットアップした。1つ目では、蛍光核染色を必要とせずに透過光画像から個々の細胞を同定できるStainFree解析を用いて、個々の細胞を同定した。同定された各セルについて、ミトコンドリア染色を表す赤色蛍光シグナルが測定され、ソフトウェアの出力パラメータとして取り込まれた。
StainFreeで得られた結果が核染色で得られた結果と同等であることを確認するために、2つ目の解析が行われた。ここでは、蛍光染色した細胞核を緑色蛍光イメージ チャンネルで同定した。その結果、核識別マスクにはミトコンドリア蛍光染色が起こる細胞質領域が含まれないため、マスクを4ピクセル(7.5μm)外側に拡張または「成長」させた。これにより、赤色ミトコンドリア蛍光を定量化し、核周辺の細胞質領域についてソフトウェアで報告することが可能になった。
各分析ケースについて、化合物処理に対するミトコンドリア応答は、セルあたりの平均ミトコンドリア蛍光対化合物濃度としてグラフ化され、IC50値はソフトウェアによって自動的に計算された。
結果
PC12細胞は、バリノマイシンで処理した場合、濃度依存的にミトコンドリア標識の減少を示した。核蛍光色素を用いた画像解析でも、個々の細胞を同定するためのStainFreeテクノロジーを用いた画像解析でも、蛍光ミトコンドリア染色は細胞単位で定量することができ、化合物処理に対するミトコンドリア応答を評価するために用いることができた。図1では、細胞の同定にStainFree解析が用いられている。紫色のマスクは個々の細胞を示す。
図1. PC12細胞のStainFree解析
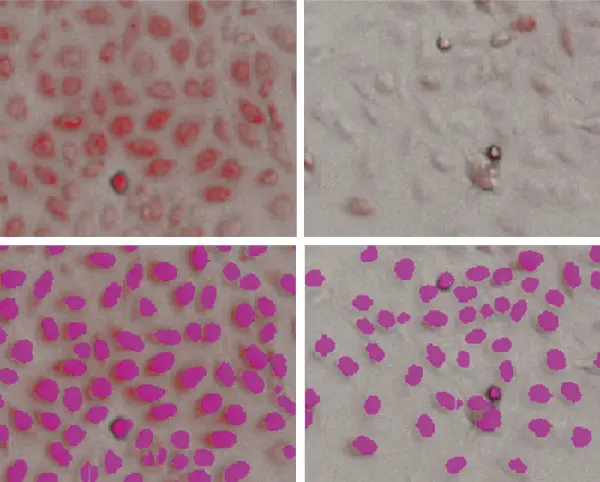
上段、透過光画像と赤色蛍光イメージャー(ミトコンドリア)を重ねたもの。下段、ソフトウェアにより識別された個々の細胞(紫色のマスク)を示すStainFree解析。左は未処理セル、右は1μMバリノマイシンで処理したセル。
図2は、個々の細胞を同定するために緑色蛍光染色した核を用いた解析である。この場合、紫色のマスクが核を示す。細胞あたりのミトコンドリアシグナルを解析するため、マスクはさらに4ピクセル拡大されている。マスクの周りの薄い境界線は、この拡張領域を示し、そのサイズはユーザーが定義できる。
図2. PC12細胞の蛍光分析
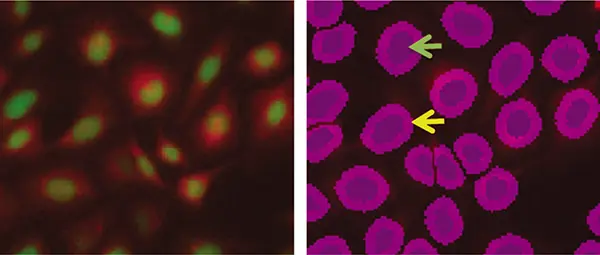
左:緑色蛍光イメージ(核)と赤色蛍光イメージ(ミトコンドリア)の重ね合わせ。右は、蛍光分析によって同定された個々の核で、濃い紫色のマスクとして示されている(緑矢印)。境界線が薄くなっているのは、対物レンズを拡張して、より多くの細胞領域を解析のために取り込んだことを示している(黄色の矢印)。
どちらの解析方法を用いても、得られるIC50値は同じです(図3)。StainFree解析は、時間と費用のかかる核のカウンター染色に代わる便利な方法です。
図3. バリノマイシンで処理したPC12細胞のIC50曲線
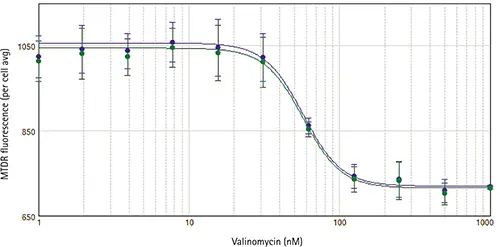
StainFree Technology(青)または蛍光核染色(緑)を用いて分析したセルについて、濃度依存性の反応曲線を示す。細胞あたりの平均ミトコンドリア蛍光は、バリノマイシンの濃度に対してグラフ化されている。どちらの解析方法でもほぼ同じ曲線が得られ、どちらの場合もIC50値は57nMであった。
結論
ミトコンドリア色素のような蛍光細胞マーカーを細胞単位で定量する必要がある場合、まず個々の細胞を同定する必要がある。その後、各細胞に関連する蛍光マーカーシグナルを定量することができる。個々の細胞を同定する方法は様々である。一つは細胞の核を蛍光色素で標識する方法であるが、これには細胞の固定が必要な場合がある。より迅速で、より安価で、より毒性の低い方法は、固定や色素を必要としないStainFree Technologyを使用することである。
このアプリケーションのハイライトでは、StainFreeテクノロジーとSpectraMax MiniMax® 300イメージングサイトメーターを使用して、まず個々の細胞を同定し、さらに化合物処理によって強度が変化するミトコンドリアマーカー染色を分析できることを示しました。その結果得られる濃度応答曲線は、核色素を使用して得られるものとほぼ同じである。StainFree テクノロジーは、DAPI のような核カウンターステインを必要とせず、固定されていないセルに対して分析を行うことを可能にします。これにより、結果が出るまでの時間が短縮され、追加実験のためにセルを生かしておくことができる、より汎用性の高いワークフローが可能になります。
以下のMolecular Devicesシステムと互換性があります
SpectraMax i3 プラットフォームと SpectraMax MiniMax 300イメージングサイトメーター
SpectraMax i3x Multi-Mode Detection Platformの詳細はこちら >>
PDF版(英語)