Application Note Organ-on-a-chipアッセイの自動化:
血管新生の培養、イメージング、解析の自動化
- アッセイのスループットの向上
- データ再現性の向上
PDF版(英語)
はじめに
Angeline Lim, Oksana Sirenko | Molecular Devices
Arthur Stok, Matthew Delport | Mimetas
Francis Enane | Beckman Coulter Life Sciences
3D細胞モデルは、2D細胞単層で培養したセルよりも、細胞の3D微小環境をよく再現できるため、多くの研究分野でますます人気が高まっている。臨床試験における化合物の高い失敗率は、2D細胞培養システムの不十分さに起因している可能性がある。そのため、創薬においてより優れた薬剤候補の選択を可能にするため、より生理学的に適切なモデルが求められている。
細胞バイオロジーにおける新しい手法と生物工学の革新とが相まって、3D培養の利用が拡大している。これらの3D細胞モデルには、単純なスフェロイドから、オルガノイドやOrgan-on-a-chip(OoC)システムのような、より複雑な構造まで含まれる。3Dモデルの複雑化が、研究や薬剤スクリーニングに広く採用されるためのハードルとなっている。具体的には、均一な3D構造の作製能力、再現性のある細胞播種、最終的にはハイスループット・アプリケーションへの3D培養の適応などが課題として挙げられる。細胞培養プロセス、エンドポイントアッセイ、解析の自動化は、3D細胞モデルの使用を促進し、スケールアップするために必要なツールを提供することができる。
3D細胞培養に関連するいくつかの課題に対処するために、我々はOrgan-on-a-chipモデルの自動化法を開発した。この自動化法は、細胞培養の自動化とモニタリングに適合した複数のインストゥルメンテーションからなる統合ワークセルを利用する。概念実証のために、OrganoPlate® 3-lane 64(MIMETAS社製)を用いて血管新生モデルを作成した。このOrganoPlateタイプは、384ウェルイメージングクオリティープレートフォーマットの64マイクロ流体ユニットで構成されており、自動リキッドハンドリングとハイコンテントイメージングに非常に適している。細胞外マトリックス(ECM)の分注と内皮細胞の播種はリキッドハンドラーで行った。Wave機能を備えた自動化対応LiCONiCインキュベーターは、マイクロ流路の連続灌流を可能にした。長時間の細胞培養のために、培地交換のようなプロセスは自動化ソフトウェアでスケジュールすることができる。エンドポイントアッセイの出力には、3Dにおける血管新生の定量化のためにハイコンテントイメージャーと解析が用いられた。細胞播種、培地交換の自動化、および3D血管系成長の定量化モニタリングのためにここで開発された方法は、化合物スクリーニングや毒性研究など、他のアプリケーションにさらに実装することができる。
方法
オルガノプレート3レーンにおける3D血管新生モデル
血管新生モデルの構築には、OrganoPlateと呼ばれるハイスループットのマイクロ流路プラットフォームを用いた。OrganoPlate 3-laneは64個または40個の組織培養チップで構成され、それぞれが3つのチャンネルで構成されている(図1)。コラーゲンI細胞外マトリックス(ECM)ゲルは各チップの中央のチャンネルに播種され、フェーズガイドによってパターニングされた。内皮細胞(初代、細胞株、iPSC由来)は各チップの右チャンネルで増殖させ、灌流をシミュレートするため、一定間隔でプレートを揺らして重力で培地が流れるようにプログラムされたLiCONiC WaveインキュベーターにOrganoPlateをプレーティングして、灌流下で内皮血管を形成させた。左側のチャンネルに血管新生因子のカクテルを添加すると、親血管から血管新生スプラウトが誘導形成された。血管新生スプラウトは1-5日間形成させた後、定量的比較のために固定し染色した。血管セルとスプラウトは4%ホルムアルデヒドで固定し、VE-カドヘリンに対する一次抗体、次いでAlexa488二次抗体(緑)で染色し、核はHoechst(青)で染色した。
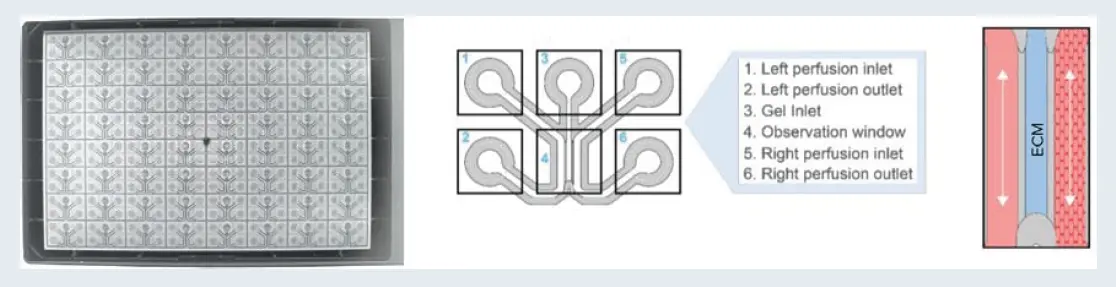
図1. OrganoPlate 3-laneは64個または40個の培養チップで構成されている。左:OrganoPlate 3レーン64の底面側から見た図。中央: OrganoPlate 3レーン64組織チップの模式図。右:ECMゲルに対して増殖した細胞の細管を示す図。

自動化のためのワークセルのセットアップ
The workcell consisted of the following primary components: Hotel for storage of microtiter plates, LiCONiC STX44 automated incubator with wave function, AquaMax Microplate Washer (Molecular Devices), HiGTM4 automated centrifuge (BioNex Solutions Inc), SpectraMax iD5 Multi-Mode Microplate Reader, ImageXpress Confocal HT.ai system, ImageXpress Pico Automated Cell Imaging System (Molecular Devices), and Biomek i7 Automated Workstation (Beckman Coulter Life Sciences). The plate handling robot is a PreciseFlex400 robot on a 2 meter rail to access all plate nests. The Green Button Go scheduling software is used to create the automation workflow and schedule setup. This software provides single user interface for control of all devices in the workcell.
OrganoPlate の自動化には、プレート調製(ECM 播種)とセル播種に Biomek i7 自動ワークステーションを使用した。Biomek i7 ワークステーションは、成長因子の添加、培地交換、細胞の固定と染色にも使用できる。OrganoPlateの移動は、培地交換のためにPreciseFlex400ロボットでBiomek i7リキッドハンドラーに行うことができる。インキュベーターからイメージング装置への移動は、細胞の長期不連続モニタリングのために設定することもできる。
結果
細胞培養工程は通常、手動での作業であり、複数のポイントでユーザーの介入が必要である。これらの工程は、統合ワークセルを用いて自動化することができる(図2)。例えば、培地交換のためにマイクロプレートをインキュベーターから無菌リキッドハンドラーに移すことができる。さらに、化合物処理とエンドポイントアッセイも同様に自動化できる。細胞培養の維持管理(モニタリングと給餌)、エンドポイントアッセイを自動化することで、再現性のある結果が得られ、リソースを節約でき、拡張可能な自動化が可能である。
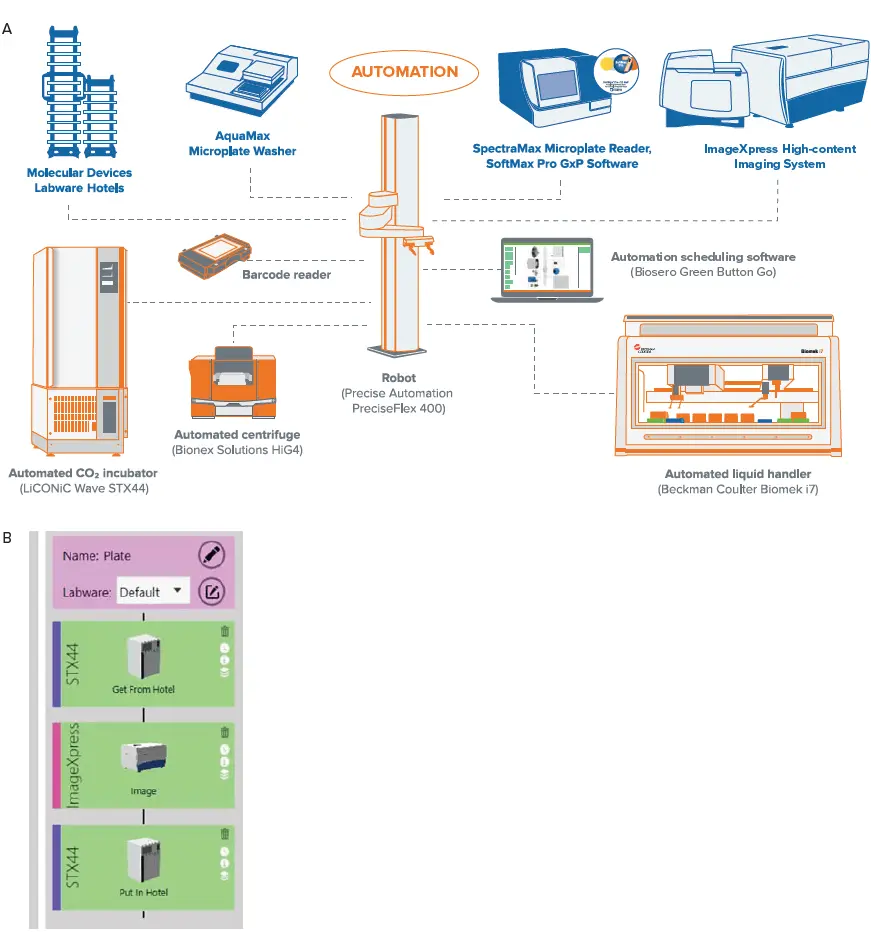
図2. ワークセル内の各インストゥルメンテーションのレイアウトを(A)に示す。インストゥルメンテーションは統合ソフトウエア(Green Button Go)によって制御され、プロセスのセットアップが可能である。培養中のセルをモニターするプロセスの例を(B)に示す。ここでは、プレートをインキュベーターからImageXpress® Confocal HT.aiに移動して明視野でイメージングし、その後インキュベーターに戻している。このプロセスはスケジューリングすることもでき、イメージングが必要なプレートをリストとして入力することで、バッチ処理を簡単に行うことができる。培地交換(フィーディング)のためのリキッドハンドラーを含む、より複雑なルーチンを実装することもできる。
拡張可能なマイクロ流路プラットフォームを用いた血管新生モデルの自動化
OoC技術は、生理的臓器とその環境を模倣するのに成功裏に使用されてきた。OoCモデルは、組織-器官相互作用、セルパターニング、組織境界、濃度勾配など、組織微小環境に関連する主要な生理学的条件の制御を可能にする。
ここでは、OrganoPlate 3-lane 64(MIMETAS)を用いて、自動化されたプラットフォーム上で血管新生萌芽をin vitroでモデル化する(図3)。OrganoPlateは灌流と濃度勾配を統合し、血管新生萌芽と微小血管安定化の研究に使用できる。リキッドハンドリングを自動化することで、アッセイの再現性が向上し、スループットが向上する。
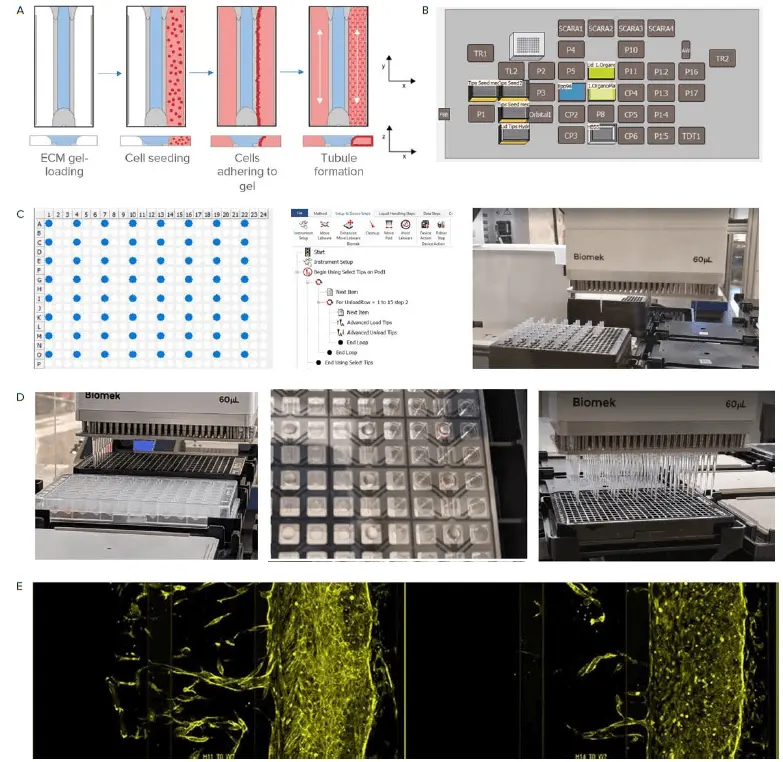
図3. 自動リキッドハンドラーによるサンプル調製。A) OrganoPlateの準備手順。観察ウィンドウを示す。青色はゲル注入口から注入されるECM。次に、ECMチャンネルに隣接する灌流チャンネルにセルを播種する。OrganoPlateは、細胞がゲルに対して沈降して接着するような角度で置かれる。最後に、培地灌流を開始し細管形成を可能にするため、プレートをロッカープラットフォーム(または波動機能付きLiCONiCインキュベーター)に置く。B) Biomek i7リキッドハンドラーには、6つのヒータークーラーサーモブロック(CP)が装備されている。ECMはこれらのCPの位置で冷却される。C) Biomek i7 リキッドハンドラーを使用して、OrganoPlate フォーマットを反映するようにピペットチップを配置することができる。D) 左側の画像はECMのローディングを示す。中央の画像は、ゲル注入口とゲルチャンネル内のECMを自動化を用いてゲルローディングした後を示す。右の画像は、灌流注入口への細胞培地の添加を示す。E) 時間経過に伴うOrganoPlate 3-lane 64における血管新生の芽生え。
経時的な血管新生スプラウトの形成
OrganoPlate 3-レーンを血管新生のモデルに用いた。内皮細胞を播種して細管を形成した。ECM全体に濃度勾配を作るために、反対側のチャンネルに成長因子を加えた。血管新生の芽が成長因子を封じ込めたチャンネル(下側)に向かって成長するのが観察された(図4)。
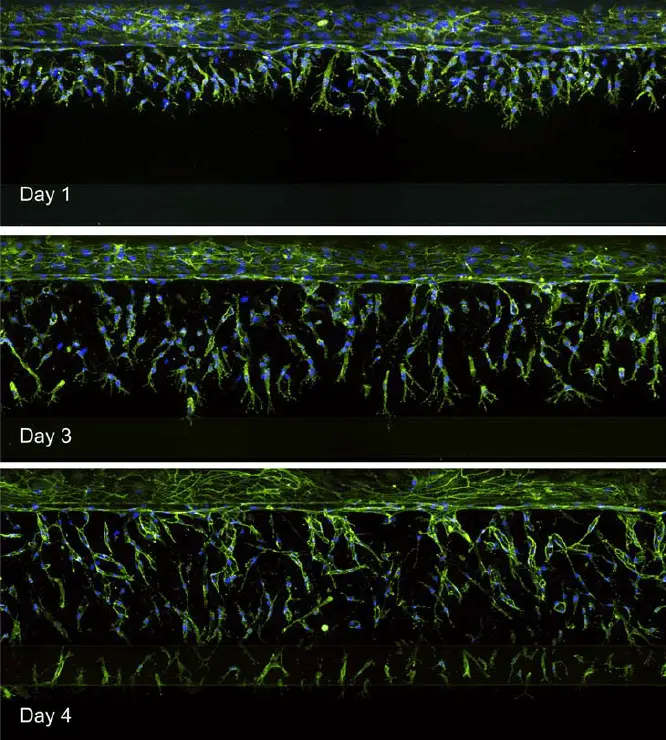
図4. 下のチャンネルに向かって成長する内皮細胞(上のチャンネル)のイメージング。経時的な血管新生萌芽の最大プロジェクション画像を示す。
血管新生萌芽の経時的定量化のための3D解析
血管新生の時間依存性を5日間にわたってモデル化した。スプラウトの数と体積の時間依存的増加が観察され、セルや核の数の増加も観察された。画像は3Dで解析され、血管新生の新芽の成長は、新芽と核の総数、それらの強度、体積、対物レンズ間の距離を含む複数の読み出しによって特徴づけられた。個々のスプラウトあたりの核の数(または平均)も決定できる(図5)。二次解析はMicrosoft Excelを用いて行った。
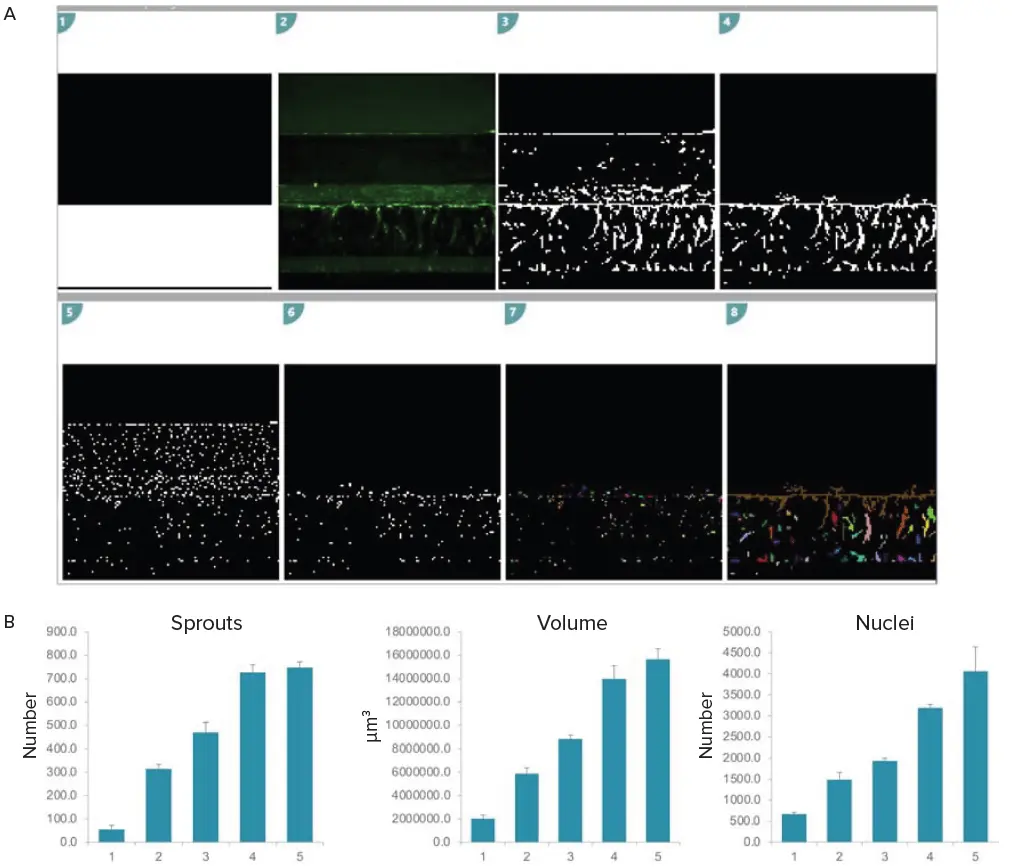
図5. 血管新生の3D解析。A) MetaXpressカスタムモジュールエディター(CME)での解析セットアップ。簡単に説明すると、ECMチャンネルを表す解析領域が作成された(白いオーバーレイ)。スプラウトのセグメンテーションを改善するために画像前処理を行った。解析領域内のスプラウトのみを定量化した(1-4)。同様のセットアップを核の定量化にも用いた(5-6)。このセグメンテーションは、すべてのz平面上で繰り返された。得られたセグメンテーションマスクを結合して、体積測定値を作成した(7-8)。B) 3Dにおけるスプラウトの定量的成長を経時的に表した棒グラフ。アッセイはトリプリケートで行い、エラーバーはSTDEVを表す。
結論
- 我々は血管新生のOoCモデルの自動ワークフローを開発した。
- 自動化の利点には、アッセイのスループットを向上させる能力とデータの再現性の向上が含まれる。
- 血管新生の3D画像と解析は、複雑な生物学的プロセスを記述する定量的指標を提供する。
PDF版(英語)