Application Note THUNDER TR-FRETアッセイによる
ERK1/2リン酸化の細胞ベース測定
- シンプルで迅速なアッセイワークフローを備えた、非常にロバストなホモジニアス(無洗浄)アッセイ。
- 細胞を血清飢餓状態にすることなく、明確で完全な濃度反応曲線を生成する高感度アッセイ。
- EC50とIC50の値は公表データと一致
- 学術および創薬アプリケーションの両方に適している
PDF版(英語)
はじめに
Joyce Itatani | Applications Scientist | Molecular Devices
Cathy Olsen, PhD | Sr. Applications Scientist | Molecular Devices
MAPK/ERK細胞シグナリング経路は、細胞表面に位置する受容体チロシンキナーゼの活性化から始まり、一連のキナーゼリン酸化ステップ(キナーゼカスケード)を経て、遺伝子発現の変化をもたらす転写因子の活性化へと至る。ERK1/2とも呼ばれるERK1とERK2は、この経路の一部としてリン酸化され、細胞増殖と分化に関与する転写因子を含む他のオンターゲットをリン酸化し活性化する。この経路の破壊や調節異常は、様々ながんの発症につながる可能性がある1。
THUNDER™は、Bioauxilium Research社が開発した、汎用性が高く費用対効果の高い細胞ベースアッセイプラットフォームであり、学術研究と創薬研究の両方に適合する方法で、リン酸化された個々の内因性タンパク質と総内因性タンパク質を測定することができる。THUNDER™ Phospho-ERK1/2 (T202/Y204)アッセイキットは、均一性時間分解フェルスター共鳴エネルギー移動(TR-FRET)サンドイッチイムノアッセイであり、3つのステップからなるシンプルなワークフローを採用している(図1)。細胞処理後、細胞を溶解し、溶解液中のPhospho-ERK1/2 (T202/Y204) を一対の蛍光標識抗体を用いて、洗浄ステップのないシングルステップの試薬添加で検出する。第一抗体Eu-Ab1は、ドナー蛍光体として機能するユーロピウムキレートで標識されている。第二の抗体 FR-Ab2 は遠赤アクセプター蛍光体で標識されている。

***図1.
THUNDER TR-FRETのワークフローは3ステップで構成され、洗浄は行わない。2プレート(トランスファー)法を示す。
標的ERK1/2タンパク質上の異なるエピトープにこれら2つの標識抗体を結合させると、ドナーとアクセプター蛍光体が近接し、ドナーのユーロピウムキレートの励起がアクセプター蛍光体へのFRETを引き起こし、665 nmにTR-FRETシグナルが生じる(図2)。ユーロピウムキレートからの残留エネルギーが615 nmの光を発生する。665 nmのシグナルは細胞溶解液中のPhospho-ERK1/2 (T202/ Y204)の量に比例し、データは665 nmのシグナルまたは665 nm/615 nm比のいずれかで表される。
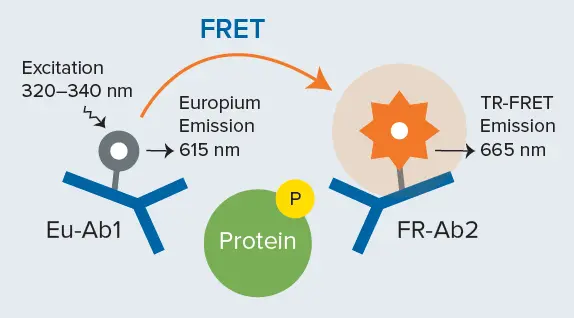
***図2.*.
THUNDER TR-FRETサンドイッチイムノアッセイ。Eu-Ab1とFR-Ab2の両方が分析物に結合することで、ユーロピウムキレートからアクセプター蛍光体へのエネルギー移動が可能となり、時間分解検出モードのマイクロプレートリーダーを用いて検出される665nmのシグナルが得られる。
ここでは、費用対効果の高いTHUNDER Phospho-ERK1/2(T202/Y204)アッセイを用いて、HEK293細胞におけるEGF誘導性のERK1/2リン酸化を測定できることを示す。また、非小細胞肺がんや膵臓がん2 の治療に用いられる薬剤であるエルロチニブ(タルセバ®)によるこのリン酸化の阻害を評価する方法も示す。SpectraMax® iD5、i3x、およびM5eマルチモードマイクロプレートリーダーとSoftMax® Proソフトウェアを使用すると、合理的なデータ解析とグラフ化により、堅牢なアッセイデータが生成されます。
材料
- THUNDER Phospho-ERK1/2 (T202/Y204) TR-FRET 細胞シグナリングアッセイキット (Bioauxilium cat #KIT-ERKP-100)
- 96ウェルクリア、ポリ-L-リジンコートマイクロプレート (Corning cat. #354516)
- 白色96ウェルハーフエリアマイクロプレート (Corning cat. #3693)
- HEK293 細胞株 (ATCC cat. #CRL-1573)
- 増殖培地 フェノールレッド(Corning cat. #10-010-CV)+10%ウシ胎児血清(FBS, Avantor Seradigm cat. #1500-500)+1%ペニシリン/ストレプトマイシン(Thermo Fisher cat. #15070)を封入したMEM(Minimum Essential Medium)。
- リコンビナントヒトEGF(R&Dシステムズ社、cat. #236-EG)
- エルロチニブ塩酸塩(Sigma cat.)
- 水、分子生物学グレード(Growcells 社、cat. #NUPW-0050)
- SpectraMax iD5 マルチモードマイクロプレートリーダー(Molecular Devices cat:
⚪︎改良TRFモジュール
⚪︎フィルターリング 320 nm BW 100 nm、616 nm BW 10 nm、665 nm BW 10 nm - SpectraMax i3xマルチモードマイクロプレートリーダー(Molecular Devices社カタログ番号i3x)
⚪︎TR-FRET検出カートリッジ(HTRF®、Molecular Devices P/N 0200-7011) - pectraMax M5eマルチモードマイクロプレートリーダー(Molecular Devices社製品番号#M5e)
メソッド
セル調製
増殖培地(MEM/10% FBS/1% penicillin/streptomycin)中のHEK293細胞を、96ウェルポリ-L-リジンコートマイクロプレートに50,000細胞/ウェル、50μL/ウェルで播種した。セルは37℃、5% CO2で一晩接着させ、増殖させた。血清飢餓は必要なかった。
試薬の調製
- 5X Lysis Buffer 1を超純水で1Xに希釈し、最終濃度1Xに希釈した100X Phosphatase Inhibitor Cocktailを加えることにより、1X Supplemented Lysis Bufferを調製した。補足1X溶解バッファー1は、1mMフッ化ナトリウム、2mMオルトバナジン酸ナトリウム、2mMβ-グリセロリン酸塩を封じ込めた。
- 1X Detection Buffer は、10X Detection Buffer を超純水で希釈して作製した。
- 4X Antibody Detection Mix in 1X Detection Buffer は、ユーザーマニュアルの指示に従い作製した。100 点キットの場合、255 μL の 1X Detection Buffer を 5 μL の Eu-Ab1 原液の入ったバイアルに加えました。20μLのFR-Ab2ストック溶液の入ったバイアルに240μLの1X Detection Bufferを加えた。260μLの希釈済みEu-Ab1と260μLの希釈済みFR-Ab2を合わせ、穏やかに混合した。
EGF刺激
10 nMから0.003 nMまでのEGFの2X作業溶液50 μLを、二重ウェル中のセルに添加し、細胞を室温で10分間インキュベートした。その後、細胞層を乱さないように培地を静かに吸引した。50μLの1X補足溶解バッファー をウェルに加え、環状シェーカーで30分間室温でインキュベートした。
インキュベーション後、各ウェルの細胞溶解液15μLを白色96ウェルハーフエリアマイクロプレートに移した。4X Antibody Detection Mix 5μLを各ウェルに加え、プレートをプレートシールで覆い、室温で4時間インキュベートした。その後、プレートを SpectraMax iD5、 SpectraMax i3x、 SpectraMax M5e リーダーで表 1 の設定を用いて読み取った。
| パラメータ | iD5設定 | i3x設定 | M5e設定 |
|---|---|---|---|
| 光学構成 | (N/A) | カートリッジ - HTRF | (N/A) |
| リードモード | TR-FRET (改良TRFモジュールとフィルターの取り付けが必要です。) |
TR-FRET | TRF |
| 読み取りタイプ | エンドポイント | エンドポイント | エンドポイント |
| 波長について |
フィルタを使用する 励起 320 nm 発光1: 616 nm 発光2: 665 nm |
励起: 340 nm 発光1: 616 nm 発光2: 665 nm |
励起: 314 nm 発光1: 616 (カットオフ 570 nm) 発光2: 665 (カットオフ630 nm) |
| プレーティングタイプ | 96 ウェル コーニング ハーフエリア | 96 ウェル コーニング ハーフエリア | 96 ウェル コーニング ハーフエリア |
| PMTと光学系 |
読み取りあたりのフラッシュ数:100 励起時間: 0.05 ms 測定遅延時間 0.05 ms 積分時間:0.3 ms 読み取り高さ: 9.31 mm |
パルス数:100 励起時間: 0.05 ms 測定遅延時間: 0.05 ms 0.05 ms 積分時間:0.3 ms 読み取り高さ: 6.28 mm |
読み取りごとのフラッシュ数: 100 測定遅延時間 0.1 ms 積分時間: 0.3 ms |
| その他の設定 | 読み取り前最適化オプションの表示 | 読み取り前最適化オプションの表示 |
*320 nm Exフィルターを使用すると、SpectraMax iD5リーダーで最適な結果が得られますが、標準的な340 nmフィルターを使用することもできます。
表 1. SpectraMax iD5、i3x、M5eリーダーの推奨設定。SpectraMax iD5およびi3xリーダーの場合、マイクロプレートおよびリードハイトは、使用するマイクロプレートの新しいロットまたはアッセイ量ごとに最適化する必要があります(SoftMax Proソフトウェアの装置設定でShow Pre-Read Optimizationオプションを使用)。
エルロチニブ阻害
25μLのエルロチニブ4倍希釈液をセルに添加し、最終濃度を1:3連続希釈で10μM~0.001μMとした。プレートを室温で15分間インキュベートした。次にセルを、上記のEGF刺激アッセイに記載されたように決定 されたEC80 0.7 nMのEGFで刺激した。EC80(2.8nM)のEGFの4倍作用溶液25μLをウェルに加え、プレ ートを室温で10分間インキュベートした。培地を注意深く除去し、50μLの1X補充溶解バッファー を各ウェルに加えた。その後、プレートを室温で30分間、環状シェーカーでインキュベートした。
インキュベーション後、各ウェルの細胞溶解液15μLを白色96ウェルハーフエリアマイクロプレートに移した。4X Antibody Detection Mix 5μLを各ウェルに加え、プレートをプレートシーラーで覆い、室温で4時間インキュベートした。その後、プレートをSpectraMax iD5、SpectraMax i3x、SpectraMax M5eリーダーで表1の設定を用いて読み取った。
データ解析
TR-FRET比は、665nmのRFU値を616nmのRFU値で割って算出し、得られた数値に1000を乗じた。バックグラウンドサブトラクションは必要なかった。TRFRET比を、SoftMax Proソフトウェアの4パラメータロジスティック方程式を用いて、EGF濃度(刺激アッセイ用)またはエルロチニブ濃度(阻害アッセイ用)に対してプロットした。得られた曲線から、EC50 値と IC50 値がソフトウ ェアによって自動的に算出された。
結果
HEK293細胞のEGF刺激により、ERK1/2(T202/Y204)リン酸化が増加し、その濃度応答はシグモイド曲線に従った。TR-FRET比を4パラメータロジスティックカーブフィットを用いてEGF濃度に対してプロットし、そこからSpectraMaxリーダーで得られたデータについて0.28 nMから0.41 nMのEC50値を算出した(図3)。これらの値は、公表されているTHUNDER TR-FRETアッセイのEC50値と一致していた3。
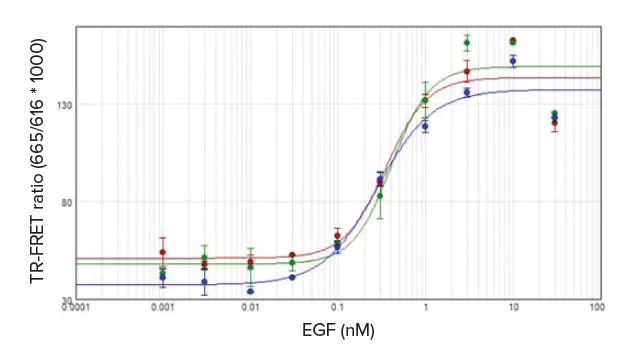
***図3.
EGFによって誘導される濃度依存的なERK1/2(T202/Y204)リン酸化。SpectraMax iD5(赤)、i3x(緑)、M5e(青)リーダーで得られた結果を4パラメータロジスティックを使ってプロットし、SoftMax Proソフトウェアを用いて0.28 nM~0.41 nMのEC50値を算出した。
注:THUNDERアッセイでは、SpectraMax M5eリーダーの性能はSpectraMax M5リーダーの性能と同等である。
エルロチニブによるリン酸化阻害の測定能力は、細胞を濃 度を増加させたエルロチニブでインキュベートした後、最初の 刺激アッセイから決定されたEC80濃度である0.7 nM EGFで細胞を 刺激することにより試験された。各SpectraMaxリーダーについて、SoftMax Proソフトウェアで得られた4パラメータカーブフィット処理から、42 nMから63 nMのIC50値が算出された(図4)。ここでも、以前に発表された値3 と一致した。
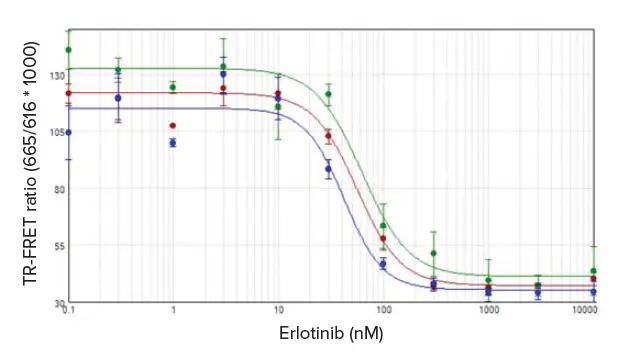
***図4.*.
エルロチニブによるEGF誘発ERK1/2リン酸化(T202/Y204)の阻害。SpectraMax iD5(赤)、i3x(緑)、M5e(青)リーダーで得られたデータを SoftMax® Proソフトウェアの4パラメータロジスティックを使ってプロットし、曲線から42~63 nMのIC50値を算出した。
|
EC 50 (EGF) |
アッセイS/B(EGF) |
IC 50 (エルロチニブ) |
アッセイS/B(EGF) | |
|---|---|---|---|---|
| SpectraMax iD5リーダー | 0.35 nM | 3.4 | 56 nM | 3.7 |
| SpectraMax i3xリーダー | 0.41 nM | 3.7 | 63 nM | 3.8 |
| SpectraMax M5eリーダー | 0.28 nM | 4.5 | 42 nM | 3.8 |
表 2. THUNDER Phospho-ERK1/2 (T202/Y204) TR-FRET アッセイに関するデータの要約。EGF誘導性リン酸化およびエルロチニブによるリン酸化阻害について、各リーダーで得られたEC50、IC50、およびアッセイのSB比を示す。
結論
全体として、THUNDER Phospho-ERK1/2(T202/Y204)アッセイは、SpectraMax iD5、i3x、M5eリーダーで、頑健なTR-FRETシグナル、広いダイナミックレンジ、高いS/B比、期待される薬理作用を示した。今回のデータは、Molecular Devices社のリーダーにおけるTHUNDER細胞シグナリングアッセイプラットフォームを検証するものである。
参考文献
- Braicu C, Buse M, Busuioc C, Drula R, Gulei D, Raduly L, Rusu A, Irimie A, Atanasov AG, Slaby O, Ionescu C, and Berindan Neagoe I. MAPKに関する包括的レビュー:がんにおける有望な治療ターゲット。Cancers, 11(10) (2019), p 1618.
- Bareschino MA, Schettino C, Troiani T, Martinelli E, Morgillo F, and Ciardiello F. Erlotinib in cancer treatment. Annals of Oncology, 18:6 (2007), pp.
- THUNDER™ Phospho-ERK1/2 (T202/Y204) TR-FRET 細胞シグナリングアッセイキット テクニカルデータシート:https://bioauxilium. com/product/phospho-erk1-2-t202-y204/
PDF版(英語)