Application Note GenePix 4400Aマイクロアレイスキャナーを使用して、
CGH 2.1Mフィーチャーアレイの高解像度画像を作成する
はじめに
スティーブン・ブッチャー(M.S.)、ショーン・キャリエド(Ph.D.)、モレキュラー・デバイス、サニーベール、1311オーリンズドライブ、 CA 94089.
ゲノムワイド関連研究は、完全なDNAセット、すなわちゲノムから特異性疾患の変異を迅速に同定するために用いられ、喘息、癌、糖尿病、精神疾患などの複雑な疾患を研究する研究者にとって有益な手法である。このような大規模な研究を促進するために、1枚のマイクロアレイスライドに210万(210M)プローブを搭載した高密度マイクロアレイは、遺伝子発現解析、ChIP-Chip、アレイ比較ゲノムハイブリダイゼーション(aCGH)アッセイを実施するための費用対効果の高いソリューションを科学者に提供する。Molecular Devices GenePix® 4400A マイクロアレイスキャナーは、これらの高密度アレイ内の各プローブを調べるのに必要な解像度(2.5 µm/ピクセル)を提供し、優れたプローブSB比(SNR)レベルとプローブごとに行われる蛍光コールの精度の向上を可能にします。
材料と方法
aCGHを用いたコピー数変異解析:罹患患者と非罹患対照から採取した血液サンプルからヒトゲノムDNAを抽出した。同量(34 µg)のcy5標識未罹患コントロールgDNAとcy3標識罹患患者gDNAの間で競合ハイブリダイゼーションを行った。メーカー推奨のプロトコール(NimbleGen Arrays User's Guide: CGH Analysis v5.1)を用いて、サンプルをRoche NimbleGen High Density 2.1M HD2 Arrays上で2-3日間ハイブリダイズさせた。その後、アレイを洗浄、乾燥し、GenePix 4400A Microarray Scannerでスキャンし、各プローブの蛍光シグナルを解析した。
aCGHアレイのスキャン
- GenePix ® Pro 7 Softwareの "Laser Settings "ウィンドウで、"Pixel Size "を2.5 µmに設定し、"Wavelength #1 (nm): "に635を、"Wavelength #2 (nm): "に532を選択します。終了したら "OK "を選択する。
- Hardware Settings "ウィンドウで、各レーザーの "Power (%):" を100%に設定する。Wavelength #1 - 635 "がCy5フィルターに設定され、"Wavelength #2 - 532 "がCy3フィルターに設定されていることを確認する。
- マイクロアレイをスライドホルダープラットフォームに下向きに置き、スライドのバーコード側をスキャナーの前面に近づけて蓋を閉めます。
- プレビュースキャン(Preview Scan)"ボタンを選択すると、スライド全体の低解像度(20 µm/pixel)スキャンが高速で実行され、スライド表面上のアレイのサイズと向きが決定されます。
- View Scan Area "ボタンで高解像度(2.5µm/pixel)でスキャンするカバースライドのエリアを指定し、白いスキャンエリアボックスをアレイの全エリアをカバーするように調整する。
- 最終的なData Scanを作成する前に、PMTゲインの設定を手動で最適化した。スライドはまず赤色(635 nm)レーザーでスキャンされ、PMTゲインはシグナル強度が最大でSB比が最小になるように調整された。次にアレイをGreen(532nm)レーザーでスキャンし、PMTゲインを調整してHistogramタブに示すようにRedレーザースキャンのシグナル強度プロファイルに一致させた。最適化されたPMTゲイン設定を用いて、スキャンエリア全体の最終データスキャンを行った。
PMT ゲインを最適に設定した場合、Image タブでアレイの Ratio (635/532) イメージを見ると、大部分のプローブが黄色に見えるはずです。ヒストグラムタブでは、波長635と波長532の強度プロファイルが、インストゥルメンテーションのダイナミックレンジ全体にわたって同様の分布を示し、Count Ratioが0.9~1.1になるはずです。
最終的なデータスキャンが完了したら、GenePix Proソフトウェアのインターフェースウィンドウの右側にある「ファイル...」ボタンアイコンをクリックし、「画像を保存-選択したスキャンエリア」を選択して、スキャンした画像をTIFファイルとして保存します。
保存されたTIF画像は、NimbleScanソフトウェアにインポートして処理することによりさらに解析され、得られたデータファイルはSignalMapソフトウェアを用いて表示される。これらのソフトウェアパッケージは、Roche NimbleGenアレイの解析用にRoche NimbleGenから提供されています。
結果
図1は、HD2 aCGHマイクロアレイスライドの全長スキャンを、赤色のCy5画像(左)、緑色のCy3画像(中央)、およびCy5/Cy3のマージ比画像(右)に分けて示している。全長スキャン(図1)

図2は、同じHD2 aCGHマイクロアレイを従来のマイクロアレイスキャナーの5 µm/ピクセルの解像度でスキャンした場合(上段)と、GenePix 4400A Microarray Scannerの2.5 µm/ピクセルの高解像度でスキャンした場合(下段)の画像の違いを示している。画像品質(図2)

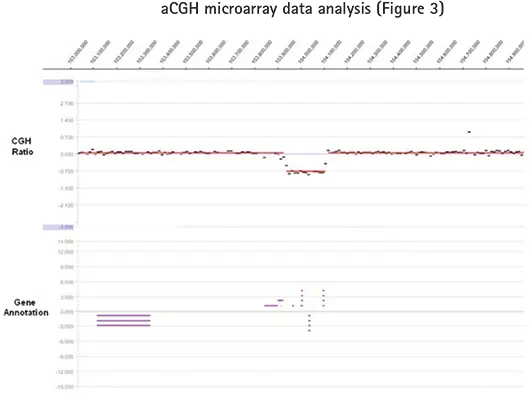
図3は、aCGHマイクロアレイをNimbleScanソフトウェアで完全に処理し、SignalMapゲノムブラウザで表示したものである。平均化されたセグメンテーションGFFファイル(デフォルトのRoche NimbleGen CGH解析設定を使用)は、患者サンプルにおけるヘテロ接合性欠失を同定する。遺伝子アノテーショントラックは、この欠失によって影響を受けるヒトゲノムの遺伝子群を同定する。
結論
GenePix 4400A マイクロアレイスキャナーは、2.5 µm/ピクセルの解像度を使用して、高密度マイクロアレイ内のプローブ境界を区別し、プローブの蛍光を正確に解析するロバスト性ツールです。ご希望の高密度アレイと組み合わせることで、研究者はコスト効率の高い方法で能力を拡張し、ゲノムワイドなアソシエイト研究をタイムリーに完了することができます。
謝辞
アイオワ大学のジョン・マナク博士に感謝したい。Manak博士は生物学部と小児科、およびRoy J. Carver Center for Genomicsの主任研究員である。このアプリケーションノートで使用したマイクロアレイデータを提供してくれたManak博士と彼の研究室の協力に非常に感謝している。
参考文献
NimbleGen Arrays User’s Guide: CGH Analysis v5.1, Roche NimbleGen, Inc.
GenePix Pro ソフトウェア v7 ユーザーガイド、モレキュラーデバイス社