Application Note ハイスループットNF-kB転写因子プロファイリング
PDF版(英語)
はじめに

転写因子は塩基配列特異的なDNA結合タンパク質であり、その遺伝子制御における重要な役割から、盛んに研究されている。転写因子核内因子-kB(NF-kB)は、感染や炎症に対する細胞応答に関与する多種多様な標的遺伝子の発現を誘導するための重要な構成要素である。NF-kBは、Relスーパーファミリーの関連タンパク質のホモ二量体およびヘテロ二量体複合体から構成されている。典型的なNF-kB複合体は、p65(RelA)タンパク質とp50タンパク質を含むヘテロ二量体で構成されている。NF-kBの活性化を正確にモニターする能力と、転写因子複合体に関与するタンパク質を同定する能力は、多くの薬剤や治療法の開発に不可欠である。最近まで、このような研究プロジェクトは時間がかかり、ハイスループットスクリーニング環境での実施は困難であった。
このアプリケーションノートでは、ImageXpress® Velos システムで測定した配列特異的 DNA 結合タンパク質の検出のために、アレイ化した二本鎖オリゴヌクレオチド(ds-DNA)からなる転写因子プロファイリングアッセイの使用について説明します。アッセイの概略図を図 1 に示す。ImageXpress Velos システムは、アレイ、ビーズ、セルベースアッセイからの蛍光測定のためのハイスループットなマルチパラメトリックスクリーニングプラットフォームです。高速スキャンにより、プレートのウェル密度に関係なく、10ミクロンの分解能で120秒/プレートのスループットが可能です。ここに示すように、このアレイ化された高度にパラレルなアッセイフォーマットは、ImageXpress Velosシステムによる高速スキャンと相まって、高スループットスクリーニング環境において、強固な感度、特異性、再現性を示す実施要件を満たしている。
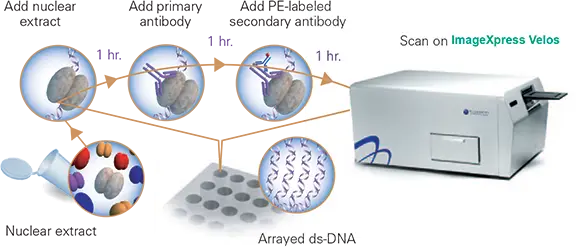
図1. アレイ化転写因子プロファイリングアッセイ概略図。
実験的
野生型kBモチーフ5'-GGGACTTTCC-3'または変異型5'-G CC ACTTTCC-3'(太字は変異部位を示す)を含むオリゴヌクレオチドは、Active Motif(カリフォルニア州カールスバッド)から入手した。片方の鎖は5'末端がアミノ修飾されていた。NF-kBのアミノ修飾ds-DNA結合配列は、Advansta社(カリフォルニア州メンローパーク)により、2x8ウェルフォーマット(96マルチウェルプレート間隔)でエポキシコーティングされたスライドグラス(Slide E、Schott-Nexterion)にピンスポットされた。各ウェルのアレイは、野生型のds-DNAと変異型kBモチーフのds-DNAの8つのスポット(直径約150mm)で構成されていた。スライドは印刷、ブロッキングされ、防湿袋(Advansta社製)に密封され、Active Motif社に出荷され、使用まで4℃で保管された。Active MotifのNF-kB TransAMTMキット(cat. #40096)のプロトコルをこれらの実験に適用した。
プロトコル
- リコンビナントp65(Active Motif、cat. # 31102)または刺激(TPA+カルシウムイオノフォア)Jurkat核抽出物(Active Motif、cat. #36013)をTransAMTM溶解バッファー(20ml)で希釈したサンプルを、30mlの結合バッファーが入った各ウェルに加える。シェーカー上で1時間インキュベートする。
- 洗浄バッファーで3回洗浄する。
- ウサギ抗p65抗体(Active Motif, NF-kB TransAMTM kit, cat.#40096)の1:1000希釈液を含む結合バッファー100mlを各ウェルに加え、RTで1時間振とうしながらインキュベートする。
- 洗浄バッファーで3回洗浄する。
- 各ウェルに100ml(1:1000希釈)のフィコエリトリン(PE)標識抗ウサギIgG(Molecular Probes, Eugene, OR)を添加し、スライドをアルミホイルで覆った状態でRTで1時間インキュベートする。
- 洗浄バッファーで4回洗浄する。
- スライドチャンバーのガスケットを分解し、スライドを斜めに持ち、洗浄バッファーで1回すすぐ。
- スライドを50mlのプラスチック製コニカルチューブに入れ、スピンドライした後、488nmレーザーで構成されたImageXpress Velos Systemでスキャンする。蛍光シグナルは540-610nmのバンドパスフィルターでフィルターした。
データ取得のためのすべての装置パラメーターは、単一の「Method」ファイルによって定義された。BlueImageTM解析ソフトウェアを用いて、8つのスポットからの平均蛍光強度を測定することにより、野生型および変異型dsDNAへのp65結合の程度を定量化した。配列したds-DNAへのp65結合による蛍光シグナルの出現を図2に示す。
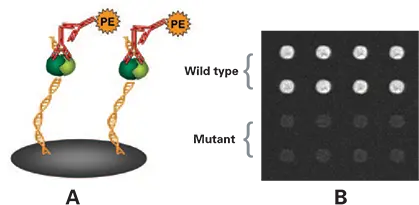
図2. A. アッセイ概略図。B. ImageXpress Velos Systemの画像。野生型ds-DNA(上部8スポット)に対するリコンビナントp65(1 ng/ウェル)の結合を示し、変異型ds-DNA(下部8スポット)に対する結合は低い。
アッセイ感度、特異性、再現性
モデルアッセイ形式は、p65(RelA)NF-kBファミリーメンバーの検出のために、各スライド上の16ウェルに印刷されたds-DNAの同じアレイで構成されていた。ds-DNAアレイに対する組換えp65の結合感度を示す標準曲線は、図3Aに示すように、タンパク質の1:2希釈系列を行うことによって作成した。アッセイの感度、特異性、バックグランドを示すため、データはバックグランドを引いていない。バックグラウンドの標準偏差の3倍に相当するシグナル-バックグラウンドとして測定されたアッセイの検出限界は、◎~0.1 ng/ウェルであった。
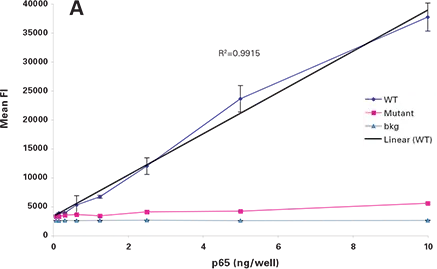
図3A. 野生型または変異型ds-DNAに対する組換え型p65の結合の標準曲線。
図3Bは、TPAおよびカルシウムイオノフォアで刺激したJurkat T細胞から調製した核抽出液中のp65の検出量の増加を示している。このデータは、このアッセイが細胞核抽出物中のp65の量に対して直線的な反応を示し、低いバックグラウンドで高いシグナル-ノイズ比を示す配列特異性を示すことを示している。アッセイの再現性を評価するために、14ウェルを5ng/ウェルのp65でインキュベートした。アッセイの再現性は非常に高く、平均±標準偏差(SD)は13,132±537蛍光強度(FI)単位で、CVは4%であった。
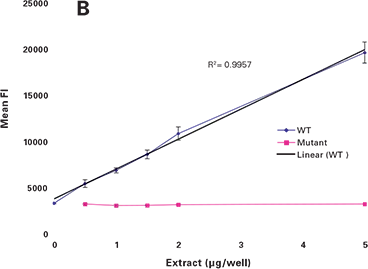
図3B. 刺激核抽出物(TPAおよびイオノフォア処理Jurkat細胞)の増加量におけるp65の定量。
結論
ImageXpress Velos Systemを使用することで、96ウェルまたは384ウェルエポキシコーティングガラスプレート(Schott-Nexterion)において、各スライド上の複数のウェルにまたがる複数の転写因子結合部位の迅速なデータ取得と解析が可能となり、これまで利用可能であった方法と比較して、より高いスループットが得られる。ここに示したデータは特にp65(RelA) NF-kBファミリーメンバーに関するものであるが、単に目的の転写因子結合部位を持つdsDNAを印刷するだけで、多数の異なる転写因子を別々に、あるいは各ウェルでスクリーニングすることができる。まとめると、ImageXpress Velos Systemは迅速なスクリーニングに適している:
- 核抽出物中の転写因子の測定
- 新規人工転写因子のコンセンサスDNA結合部位のプロファイリング
- 配列特異的DNA結合タンパク質の低分子阻害剤のスクリーニング
ハイスループットなスクリーニング環境において、あらゆる転写因子の組み合わせの研究に必要な内容でデザインされたカスタムアッセイが可能になりました。詳しくはお問い合わせください (info@moldev.com)。
PDF版(英語)