Application Note IN Carta SINAPアプリケーションモジュールによる
標識不要の細胞セグメンテーション
- 標識不要細胞のロバストなセグメンテーションにSINAPディープラーニングアプリケーションモジュールを使用
- SINAPで事前に学習させたディープラーニングモデルを画像用にカスタマイズする簡単なワークフローを利用
- SINAPの標識不要細胞セグメンテーションで信頼性の高い定量データを得る
PDF版(英語)
はじめに
Alla Zaltsman, PhD | Software Validation Scientist | Molecular Devices
Misha Bashkurov, PhD | Product Owner | Molecular Devices
Yu-Chen Hwang, PhD | Imaging Software Product Manager | Molecular Devices
標識不要の細胞解析は、蛍光色素を使用するよりも優れた代替手段を提供する。なぜなら、科学者は、遺伝子組み換えや細胞を標識・固定するための試薬を使用することによる悪影響を受けることなく、生物学的プロセスを調べるという、ほぼ本来の条件下で生細胞を画像化することができるからである。透過光(TL)画像を使用して細胞を検出することも、特異性標識の蛍光チャンネルを最大限に利用するために効果的である。
対象オブジェクトを背景や残骸などから正確に区別する対物レンズのセグメンテーションは、画像解析ワークフローに必要なステップである。しかし、標識不要顕微鏡のためのロバストな細胞セグメンテーション法は、細胞の透明な性質、撮影条件のばらつき、低コントラスト、陰影の問題のために、非常に困難な場合があります。
このアプリケーションノートでは、IN Carta™画像解析ソフトウェアの人工知能(AI)ベースのセグメンテーションツールであるSINAPを使用し、これらの困難なTLセグメンテーションの問題に取り組みます。
SINAPセグメンテーションワークフロー
SINAPはディープラーニングアルゴリズムを利用して、画像内の対象オブジェクトを検出する。これは、対象オブジェクトと背景が手動で注釈された「グランドトゥルース」画像を使用したモデル学習によって行われる。
INカルタのSINAPモジュールには、注釈付き画像の大規模なセットでトレーニングされた特異性の生物学的構造のベースモデルが含まれています。このベースモデルは、対物レンズのセグメンテーションに直接使用することができる(図1のA)。対物レンズを出発点として、そこからさらにアルゴリズムを "ティーチング "して、サンプル中の対象オブジェクトを除外し、目的の生物学的構造を認識することもできる(図1のB)。モデルの性能を向上させるためのこのようなアルゴリズムのさらなる学習は、ユーザー注釈付き画像を使用したユーザーフレンドリーなSINAPワークフロー内で行われます。学習プロセスは反復可能であり、モデルを改善するためにさらに多くの画像に注釈を付けることができる(図1、C)。
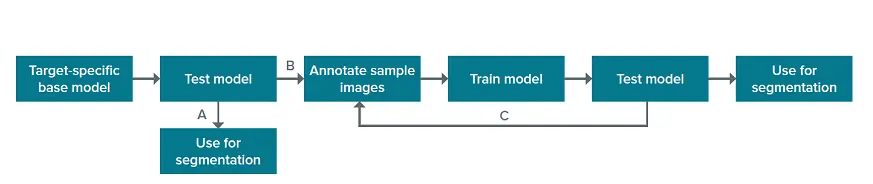
図1. SINAPセグメンテーションのワークフロー。
INカルタで提供されるベースモデルのリストには、透過光画像の細胞セグメンテーション用モデル(TL_Cellsモデル)が含まれている。
透過光セルベースモデルは標識不要の細胞セグメンテーションに使用できる。
TL_Cellsモデルは、画像特性(画像のコントラストや濃淡など)や細胞形態がトレーニングに使用した画像(図2)と類似している場合に、セグメンテーション(図1のA)に使用することができる。
図2の画像は背景にゴミが混じった凹凸があるが、このモデルは細胞の位置を特定し、背景から分離することに成功した。
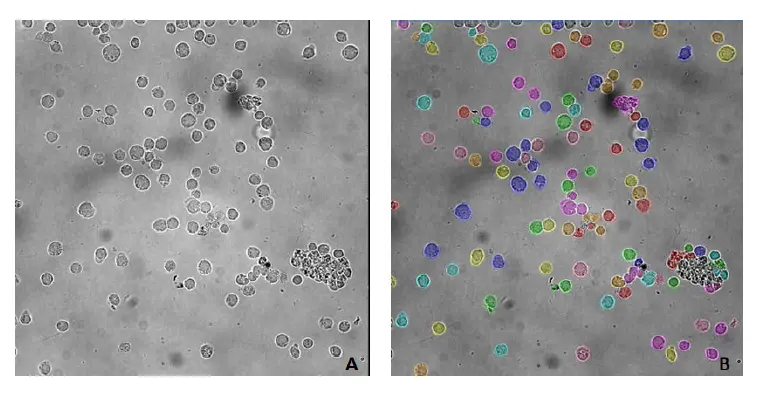
図2. TL_CellsベースモデルでセグメンテーションされたPBMC(末梢血単核細胞)。A) 元画像、B) TL_Cellsベースモデルによるセグメンテーション後のセルマスク。画像はIN Cell Analyzer 2200 (GE Healthcare)、対物レンズ40倍で取得。画像解析はIN Carta v1.15で行った。
透過光セルベースモデルは、画像内の細胞を正確にセグメンテーションするために、さらにトレーニングすることができます。
テストサンプルの注釈付き画像を追加することで、ベースモデルをさらにトレーニングし、細胞をよりよく認識し、セグメンテーション結果を向上させることができます。
SINAPモジュールには使いやすいツールが含まれており、ユーザーは画像(または関心領域)に素早く注釈を付け、トレーニングセットに追加することができます。現在のモデルが失敗している、補正された特徴を封じ込めた画像でトレーニングセットを拡張することで、モデルを微調整してわずかな改善を図ることも、より深い学習プロセスのために再トレーニングすることもできる。
必要な画像補正の程度は、図3に示すようにサンプルによって異なります。
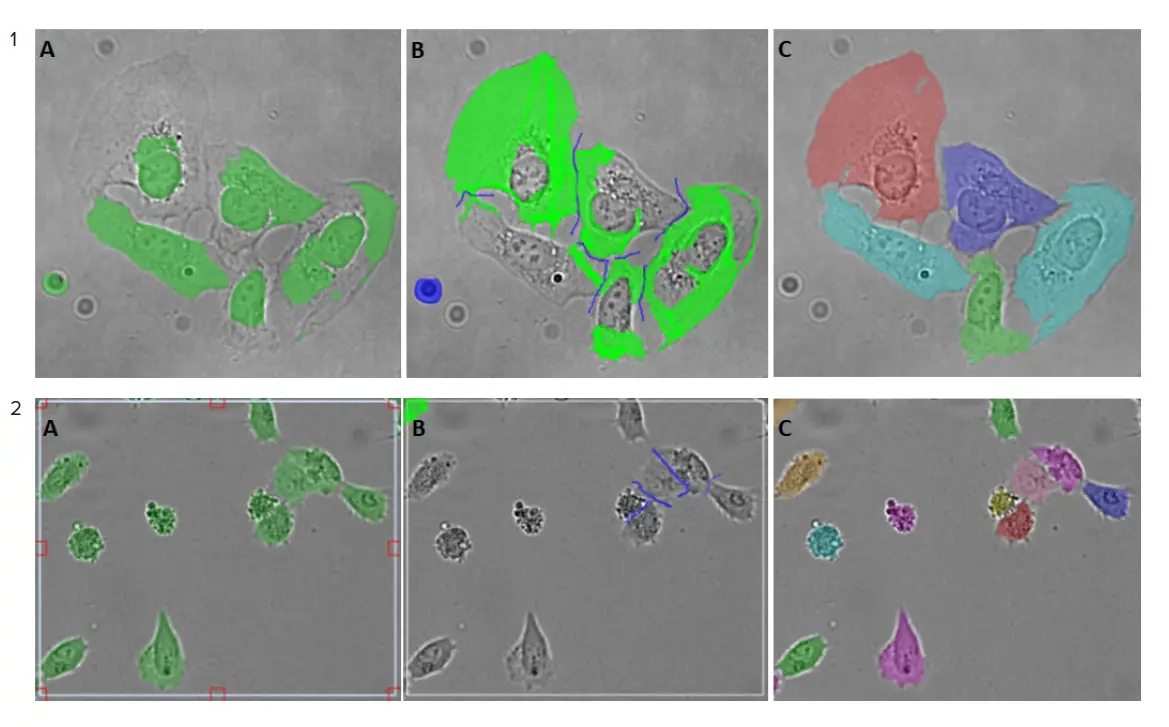
図3. 画像アノテーションプロセスの例。1) Hela細胞画像(ImageXpress® Micro Confocalハイコンテントイメージングシステムを使用、対物レンズ20倍で取得)、この例ではわずかな補正のみ必要。2) U2OS細胞の画像(IN Cell Analyzer 6000(GE Healthcare社製)を使用、対物レンズ20倍)。A-Cパネルはアノテーションプロセスのステップを表している: A) セグメンテーションされた画像から、正しくセグメンテーションされた細胞と正しくセグメンテーションされなかった細胞を含む関心領域が選択された。B) SINAPツールを用いて、細胞の輪郭を修正し(明るい緑)、背景を調整した。更新された関心領域(B)がトレーニングセットに追加された。C) トレーニングセットが収集されると、モデルがトレーニングされた。新たに学習されたモデルは画像セグメンテーションに適用された。多色のセグメンテーションマスクにより、セグメンテーションはより正確になった。
図 4 に示す例は、SINAP によるセグメンテーションのワークフ ローを示している。図 4 のパネル A では、TL-Cells のベースモデルを用いて、図 1 の A で示した SINAP セグメンテーションのワークフローに従って CHO 細胞の画像をセグメンテーションした。次に、手動で注釈を付けた関心領域(ROI)を用いてモデルを微調整した。注釈付きROIには、正しくセグメンテーションされたセルも、そうでないセルも含まれていた。モデルをトレーニングするための注釈付きROIの数を5から10に増やすと、画像内の位置に関係なく、個々の細胞をピッキングするモデルの能力が向上した(図4、パネルBとC、SINAPセグメンテーションワークフロー、図1、BとC)。

図4. SINAPモデルの微調整の例。INカルタでTL_Cellsベースモデルを用いてセグメンテーションされたCHO細胞。1) 画像全体(視野)、2) 画像のアウトライン(黄色)領域。A) TL_Cellsベースモデルによるセグメンテーション。セグメンテーションマスク(多色)は、一部の細胞しか検出されていないことを示している。B) 5つの注釈付きROIを使用して微調整したTL_Cellsモデルによるセグメンテーション。細胞の検出精度が大幅に向上した。画像はImageXpress® マイクロコンフォーカルハイコンテントイメージングシステムを用い、対物レンズ10倍で取得した。
SINAPでセグメンテーションされた標識不要細胞の数は、蛍光イメージャーでセグメンテーションされた核の数と高い相関がある。
SINAPによる細胞セグメンテーションの精度を評価するために、CHOおよびHEK 293細胞の連続希釈サンプルを用いた。核はHoechstでカウンター染色した。透過光とDAPIチャンネル蛍光イメージャーの両方を取得した。透過光画像のセルは微調整したTL_Cellsベースモデルを用いてセグメンテーションし、DAPI染色した核はNucleiロバストセグメンテーション法を用いてセグメンテーションした。
その結果(図5)、細胞の数と核の数は、細胞密度の範囲にわたってよく相関しており、ペアのt検定分析では両者に有意差は認められなかった。
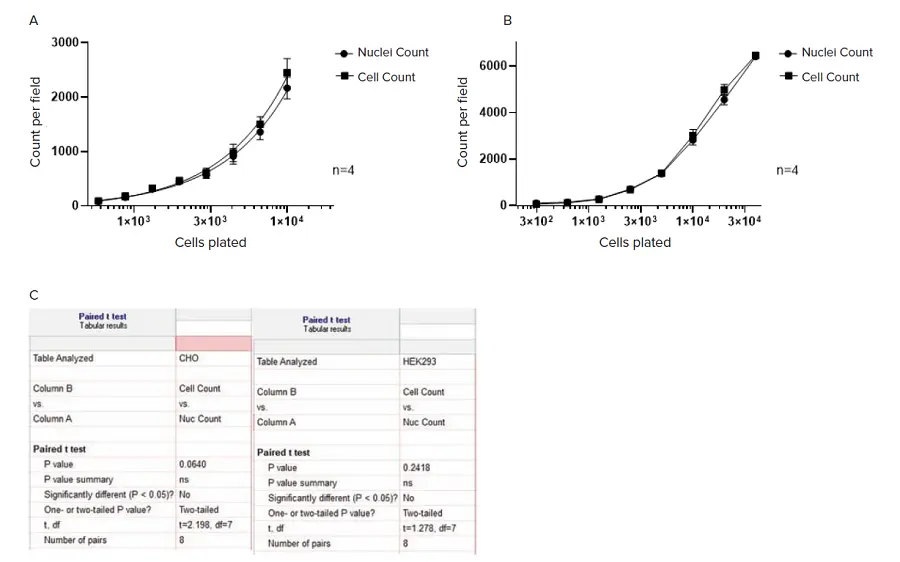
図5. SINAPでセグメンテーションされた標識不要セルと、Nuclei Robustセグメンテーションで蛍光チャンネルにセグメンテーションされた核の計数。画像セグメンテーションと解析はIN Carta画像解析ソフトウェアv1.15を用いて行った。A) CHO細胞;画像はImageXpress Microハイコンテントイメージングシステム、対物レンズ10倍で取得。B) HEK 293細胞;画像はSpectraMax® MiniMax™ 300イメージングサイトメーター、対物レンズ4倍で取得。すべてのデータは4ウェルの平均(±SD)。C) Paired t-test分析をGraphPad Prizmソフトウェアで行った。
結論
- INカルタSINAPディープラーニングモジュールは、標識不要細胞のセグメンテーションに効果的に使用できる。
- 透過光での細胞セグメンテーションのために事前に訓練されたベースモデルは、追加注釈画像による微調整や再学習によって簡単にカスタマイズすることができる。
- 標識不要の細胞セグメンテーションの精度は、透過光における核のセグメンテーションに匹敵する。
SINAPを使用した場合の蛍光チャネルにおける核のセグメンテーションに匹敵します。
PDF版(英語)