Application Note タンパク質発現と細胞増殖に対する
IPTGの影響のマルチタスクカイネティック測定
- 複数の異なる信号を同時に経時的に測定
- 包括的なデータ解析のための多様なアルゴリズムとカーブフィット処理の活用
- ワークフローエディターを使用したシンプルなプロトコルのセットアップ
PDF版(英語)
はじめに
複数の異なるシグナルアウトプットを同時に経時的に測定することは、細胞増殖や遺伝子発現に対するタンパク質や化合物の影響を調べる際に特に有用です。このアプリケーションノートでは、SoftMax® Pro 7 ソフトウェアを使用して、セル増殖(吸光度)とタンパク質発現(蛍光)を測定します。
細菌において、lacオペロンは1つのプロモーターの制御下にある遺伝子のクラスタリングであり、ß-ガラクトシダーゼをコードしています。タンパク質の塩基配列をlacオペロンを含むプラスミドにライゲーショ ンし、そのプラスミドで細菌をトランスフェクトすると、この形質転換 細菌を使って目的のタンパク質を発現させることができる。通常、lacプロモーターはアロステリックに抑制されているが、イソプロピルß-D-1-チオガラクトピラノシド(IPTG)の存在下では、プロモーター配列からリプレッサーが遊離し、目的のタンパク質が発現する。逆にIPTGが過剰になると、細胞はタンパク質発現に不釣り合いな細胞資源を費やすことになり、増殖が阻害される1。
我々は、細胞密度とタンパク質発現を同時に測定することで、大腸菌がIPTGの希釈系列にどのように反応するかを示す。
材料
- SpectraMax® M2マルチモードマイクロプレートリーダー(Molecular Devices社製)
- ルリアブロス(LB)培地(Teknova cat. #L8080)
- アンピシリンナトリウム塩(Teknova cat.#A9525)
- 1 M IPTG(Teknova 社のカタログ番号 #I3431)
- 96ウェル透明平底ポリスチレンマイクロプレート (Greiner cat. #655-161)
- pBbE5-RFPプラスミドを封じ込めた大腸菌 (Keasling Lab)
方法
pBbE5-RFPプラスミドを封じ込めた大腸菌(図1)はJay D. Keasling教授から提供された2。この大腸菌を100μMアンピシリンを封じ込めたLB培地でOD600が0.3になるまで増殖させた。その後、100 µLの大腸菌を透明な96ウェルマイクロプレートに移した。500μMから始まるIPTGの2倍連続希釈液を用いて大腸菌を処理した。0 µM IPTGと培地のみのコントロールも同様にアッセイした。
図1. pBbE5a-RFPプラスミドマップ。
次に、マイクロプレートをSpectraMax M2 マルチモードマイクロプレートリーダーにセットし、カイネティックアッセイの間、32℃でインキュベートしました。SoftMax Pro 7 Workflow Editorを使用し、吸光度測定と蛍光測定を含むカイネティック測定サイクルを表1に示す設定で作成し、測定間に10秒間のプレートシェイクを行った。カイネティックサイクルは10分ごとに1回、合計24時間繰り返すように設定した。得られたdual read modeカイネティック測定データは、SoftMax Pro 7ソフトウェアを用いて解析した。
| 読み取りモード | ||
|---|---|---|
| 吸光度 | 蛍光 | |
| 波長(nm) | 600 | Ex/Em = 530/625 |
| Read type | 下方測定 | 下方測定 |
| 読み取り間隔 | 1回/10分 | 1回/10分 |
| 光学設定 | - | 10フラッシュ/リード |
| PMTゲイン | - | 高 |
表1. 細胞密度とタンパク質発現を測定するためのインストゥルメンテーション設定。
測定結果
吸光度および蛍光測定のカイネティックトレースをそれぞれ図 2 および 3 に示す。図 2 では、IPTG 濃度を上げると OD600 値が低下し、細菌の増殖に悪影響があることが示された。図 3 では、IPTG 濃度を増加させても赤色蛍光タンパク質(RFP)の発現は増加しなかった。一方、IPTG 濃度を上げると、RFP 発現量が全体的に減少した。この RFP 発現の減少は、細菌増殖の停滞による可能性が高い。
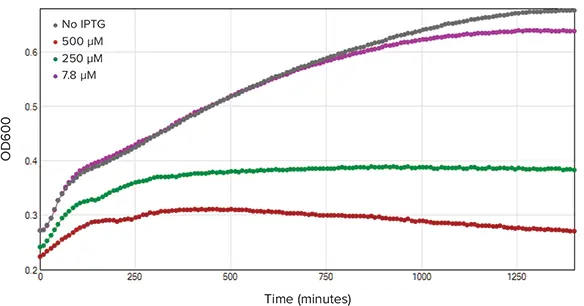
図2. OD600カイネティックのトレース。OD600の測定値は24時間にわたって10分ごとに記録した(n=3)。IPTG濃度が上昇するにつれて、細菌の増殖速度は低下した。
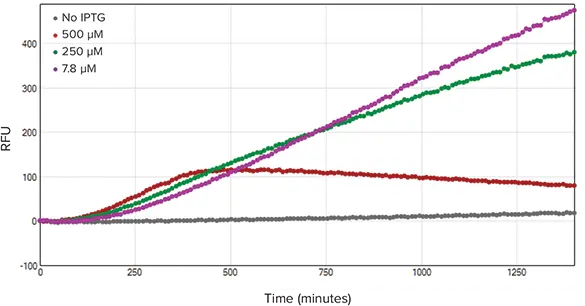
図3. 蛍光カイネティックのトレース。蛍光はOD600測定と同時に記録した(n=3)。500 µM IPTG処理ウェルを除き、蛍光出力はすべての濃度範囲で比較的類似していた。
図4では、曲線下面積(AUC)を計算するために、吸光度と 蛍光の両データセットにリダクションを適用した。この解析は SoftMax® Pro 7 ソフトウェアを用いて実施し、細胞増殖とタンパク質発現に対する IPTG の影響を要約した。蛍光に対するIPTGの影響は、IPTG濃度が高くなるにつれて細菌の増殖(吸光度)が低下した500 µMを除いて、すべての濃度で同様であった。
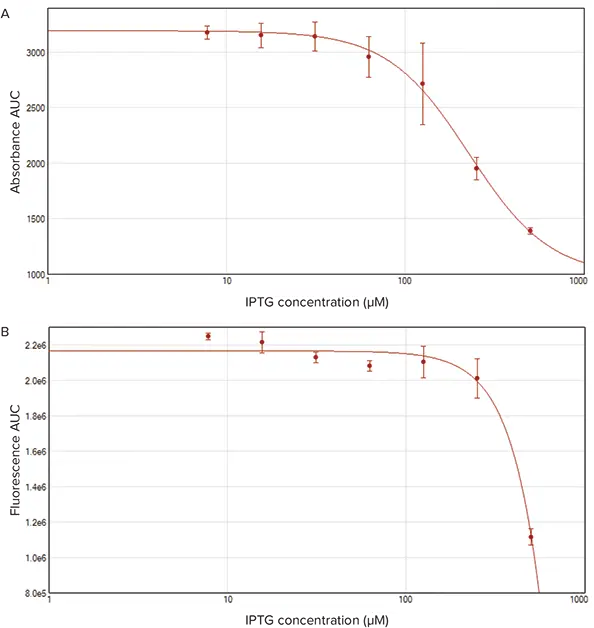
図4. 吸光度および蛍光測定の曲線下面積(AUC)。吸光度(A)と蛍光(B)のAUCは、SoftMax Pro 7ソフトウェアを用いて計算し、プロットした。細胞増殖(吸光度)およびタンパク質発現(蛍光)に対するIPTGの効果を示すために、4パラメータ曲線を適用した。
RFP/OD600値を計算することで、集団密度に対するタンパク質発現を測定することができた。SoftMax Pro 7 ソフトウェアを用いて、RFP/OD600 比を計算した(図 5)。カイネティックトレースに基づくと、250 µM IPTG処理したバクテリアがセルあたり最も多くのRFPを発現した。
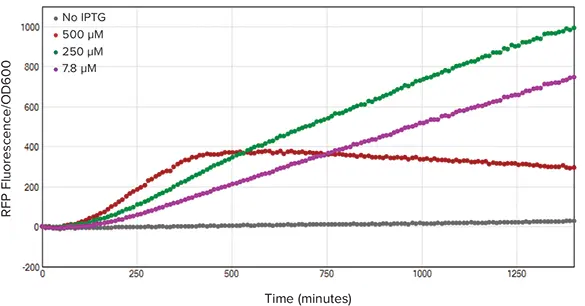
図5. RFP/OD600比。細胞密度に対する蛍光の比率は、蛍光値をOD600値で割って算出した。細胞密度に対する蛍光を正規化した結果、250 µM IPTG処理菌(緑)が細胞あたり最も高いRFP濃度を示した。
結論
Molecular Devices SoftMax Pro 7 ソフトウェアとマルチモードマイクロプレートリーダーにより、複数の生物学的事象を時間経過とともに同時に測定できることを示しました。このアプリケーションノートで示した実験コンセプトは、単純ではあるが、より複雑な実験にも応用できる。
参考文献
- Malakar, P. and Venkatesh, K.V. (2012) Lacタンパク質の発現による大腸菌のグリセロール上での増殖に対する基質およびIPTG濃度の影響。Applied Microbiology and Biotechnology, 93(6), 2543-2549.
- Lee, T.S., Krupa, R.A., Zhang, F., Hajimorad, M., Holtz, W.J., Prasad, N., Lee, S.K., and Keasling, J.D. (2011) BglBrickベクターとデータシート:遺伝子発現のための合成生物学プラットフォーム。Journal of Biological Engineering, 5, 12.
PDF版(英語)