Application Note 生細胞における
蛍光タンパク質の非侵襲的測定
- 生細胞中の蛍光タンパク質を簡単かつ非侵襲的に測定
- 個々の蛍光タンパク質に最適な結果が得られるように波長を調整
- 下方測定を行うことで最高の感度を実現
PDF版(英語)
はじめに
蛍光タンパク質は、生体内の生物学的事象をモニターするツールとして、絶大な人気を博している。クラゲのオワンクラゲ(Aequorea victoria)由来の緑色蛍光タンパク質(GFP)が最初に選択されたタンパク質であったが、現在では、強化型GFP変異体、他種のクラゲ由来の蛍光タンパク質、サンゴ礁のタンパク質など、数多くのタンパク質が利用可能である。これらのタンパク質は、バクテリアや酵母から植物や哺乳類まで、多様な細胞や生物でクローニングすることができる。蛍光タンパク質は安定で、毒性も少なく、外部補因子を必要とせずに生体内で可視蛍光を発生する能力を持つ。蛍光タンパク質は、分子タグとして、あるいは独立したレポーターとして、タンパク質の合成やターンオーバー、タンパク質のトランスロケーション、遺伝子の誘導、細胞系譜など、さまざまな細胞プロセスを可視化、追跡、定量化するために用いることができる。様々なタンパク質は色が異なるので、マルチプレックスアッセイに用いることができる。蛍光顕微鏡やフローサイトメトリーでモニターすることもできる。細胞を物理的に選別する必要がない場合や、細胞内遊走をモニターする必要がない場合、マイクロプレート蛍光分析は、より便利でハイスループットな検出システムを提供します。ここでは、Molecular Devicesマイクロプレートリーダーが、生細胞中の蛍光タンパク質を簡単かつ非侵襲的に測定できることを示す。
我々はクローンテック社から3つのHEK-293細胞株を入手し、それぞれに異なる蛍光タンパク質を安定的にトランスフェクトした。研究の目的は以下の通りである: 1)3つの細胞株に対する最適なWavelength設定を決定すること、2)検出下限(LLD)を推定するために各細胞株の希釈系列を準備すること、3)別の細胞株の存在下で1つの細胞株を識別する実現可能性を実証すること。
材料
- 蛍光タンパク質を安定に発現するHEK-293細胞株はClontech社から入手した:
◦AcGFP- Aequorea coerelescens(A.victoriaとは異なるクラゲ)からクローニングされたGFPの変異体。
◦ZsGreen-GFPに似ているが、より明るい(サンゴ礁由来)
◦DsRed- サンゴ礁からクローニングされた赤色シフト蛍光タンパク質。 - HEK-293細胞株(非トランスフェクション) (ATCC cat. #CRL-1573)
- DME:高グルコース(Irvine Scientific cat.)
- G418: Geneticin (Gibco cat. #11811-031)
- FBS (Irvine Scientific社のカタログ#3000A)
- グルタミン/ペン/Strep溶液 (Gibco cat. #10378-016)
- トリプシン/EDTA 1X in HBSS (Irvine Scientific社のカタログ#9341)
- 10X ハンク平衡塩溶液 (Gibco cat. #14065-056)
- 1M HEPES(Irvine Scientificのカタログ番号9319)
- HBSS Buffer(1X Hank's Balanced Salt Solution with 20 mM HEPES buffer): 10X Hank's Balanced Salt Solution(100 mL)、1M HEPES(20 mL)および細胞培養用水(880 mL)から調製。
- 細胞培養用水(Irvine Scientific社、商品番号9312)
- 黒壁透明底96ウェルマイクロプレート(Corning社製 cat.#3603)または黒壁透明底384ウェルマイクロプレート(Corning社製 cat.)
- SpectraMax® M5 マルチモードマイクロプレートリーダー(Molecular Devices社製)
- Gemini EMマイクロプレートリーダー(Molecular Devices社製)
方法
細胞の調製と分析
セルはDME+10%FBS+1%Pen/Strep/L-glutamine+500μg/mLのG418でバルクフラスコで培養した。トランスフェクションしていないHEK細胞はコントロールとして加えた。実験前夜、細胞をトリプシン処理し、細胞懸濁液を連続希釈して、細胞密度を50万個/mLから100個/mLにした。96ウェルおよび384ウェルのマイクロプレート(100 µL/ウェル)および25 µL/ウェルに一晩播種した。従って、播種細胞密度は50,000~10細胞/ウェル(96ウェルプレート)、12,500~2.5細胞/ウェル(384ウェルプレート)であった。96ウェルプレート、384ウェルプレートとも、1希釈につき12ウェルずつ複製した。マイクロプレートリーダーは、SpectraMax M5とGemini EMリーダーで、翌日下方と上方から読み取った。
波長の最適化
SpectraMax M5とGemini EMは、モノクロメーターベースのマイクロプレートリーダーです。これらの装置の励起および Emission 波長は、それぞれの蛍光色素のバックグラウンドに対するシグナルが最適になるように調整することができ、フィルターベースの装置よりも優れている。一般的には、励起波長を予想されるEx λmaxより20-25 nm下に設定して予備的な発光スキャンを行い、発光波長をEm λmaxより20-25 nm上に設定して励起スキャンを行う。この2つのスキャンによって、実際のExλmaxとEmλmaxの値が得られるはずである。その後、追加スキャンを行い、定量分析に最適なSB比を与える励起波長とEmission波長の組み合わせを決定する。(ストークスシフトに応じて、Ex波長を下げ、Em波長を上げ、不要な励起光を遮断するためにエミッションカットオフフィルターを使用する)。2つの異なるカットオフフィルターを選択するために、最終的なEmissionスキャンを2回行う必要がある場合もある。
結果
波長の最適化
波長最適化手順の一例として、Gemini EMリーダーでのDsRed波長スキャンを以下に示す。励起λmaxを決定するために、Emission波長を600 nmに設定して励起スキャンを行った。その結果、励起λmaxは556 nmであった(図1)。
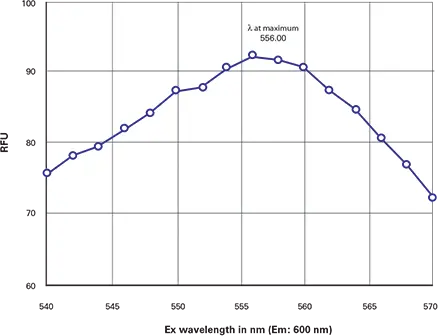
図1. DsRed励起スキャン。λmax=556nmを示すDsRedの励起スキャン。
発光λmaxを決定するために、励起波長を535に設定して発光スキャンを行ったところ、発光λmaxは584nmであることがわかった(図2)。ExとEmのλmax値は、公表値の557/579に近かった。
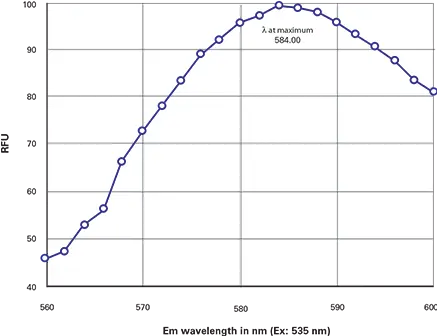
図2. DsRedの発光スキャン。DsRedの発光スキャン。λmax = 584 nmを示す。
次のステップでは、最適なシグナル/SB比を得るための最適なExとEmの設定を決定した。ストークスシフトが小さかった(22nm)ので、励起波長を下げ、Emission波長を上げて両者の分離を大きくし、さらに感度を最適にするためにEmissionカットオフフィルターを使用する必要があることは明らかであった。したがって、励起波長を550nmに下げ、570nm以上の不要な励起光を遮断するために570nmのエミッションカットフィルターを選択した。575~600 nmのエミッションスキャンを行ったところ、DsRedトランスフェクトセルでは約587~588 nmにピークが見られた(図3、上段曲線)。バックグラウンド(非トランスフェクション細胞)はその領域で比較的平坦なプロットを示した(図3、下側の曲線)。このスキャンに基づき、最適化された設定は、575 nmの Emissionカットオフフィルターを用いたEx/Em = 550/588と判断された。
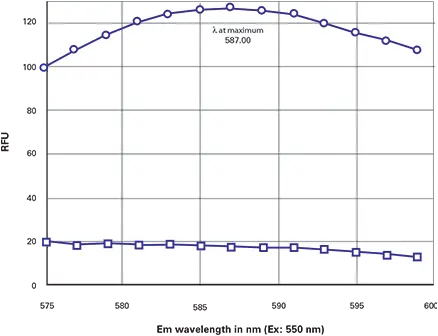
図3. DsRed設定の最適化。Emission scanは570 nmのカットオフフィルターで行い、Exは550 nmに設定した。上の曲線=トランスフェクション細胞。下はコントロール細胞。最終的に最適化された設定: Ex/Em = 550/588 + 570 Emission cutoff.
観測されたλの最大値と、定量分析のための推奨設定を表1にまとめた。
|
出版されたλ
maxima |
観測されたλ
maxima |
M5*に最適化されたセッティング | |||||
|---|---|---|---|---|---|---|---|
| 蛍光体 |
Ex λ max |
Em λ max |
Ex λ max |
Ex λ max |
Ex | Em | Cutoff |
| AcGFP | 475 | 505 | 480 | 510 | 480 | 525 | 515 |
| ZsGreen | 493 | 505 | 494 | 505 | 494 | 525 | 515 |
| DsRed | 557 | 579 | 556 | 584 | 550 | 588 |
570 |
表1. 観測されたラムダの最大値と推奨設定のまとめ。*M5とGemini EMは光学系に微妙な違いがある。Gemini EMの場合、AcGFPとZsGreenの推奨設定はEx/Em 480/510、495カットオフフィルターである。
セル希釈シリーズ
希釈系列を下方から読み取った結果を図4(96ウェル)および図5(384ウェル)に示す。上から読み取った結果を図6(96ウェル)と図7(384ウェル)に示す。ZsGreen細胞株(上の曲線)は、他の2つの細胞株より約3.5倍明るかった。DsRed細胞株はZsGreenより薄暗かったが、DsRed波長ではバックグラウ ンドが低いため、検出限界は2つの株で同程度であった(表2)。
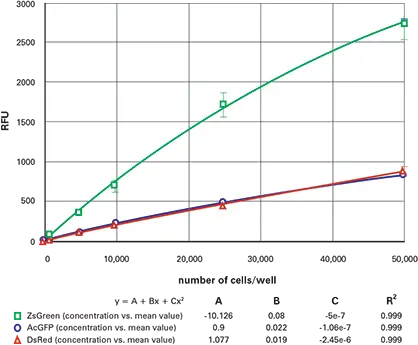
図4. 96ウェル蛍光細胞希釈系列、下方測定を行う。96ウェルプレートにおけるZsGreen(緑)、AcGFP(青)およびDsRed(赤)トランスフェクト細胞の希釈系列(下方測定を行う)。
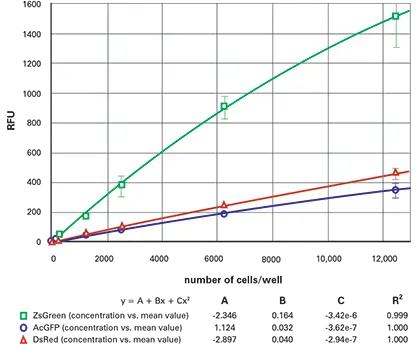
図5. 384ウェル蛍光細胞希釈系列、下方測定を行う。384ウェルプレートにおけるZsGreen(緑)、AcGFP(青)およびDsRed(赤)トランスフェクト細胞の希釈系列(下方測定を行う)。
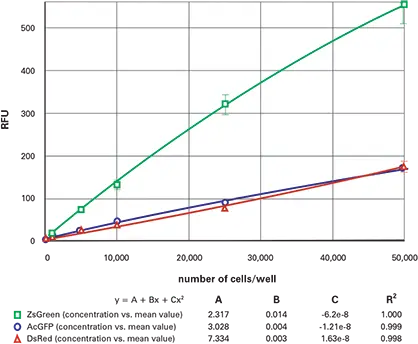
図6. 96ウェル蛍光細胞希釈系列、上から読む。96ウェルプレートにおけるZsGreen(緑)、AcGFP(青)およびDsRed(赤)トランスフェクト細胞の希釈系列、上から読む。
| タンパク質 |
下方測定を行う(#セル/ウェル)
96-Well 384-Well |
トップリード(#セル/ウェル)
96-Well 384-Well
|
||
|---|---|---|---|---|
| SpectraMax M5 | ||||
| AcGFP | 400 | 250 | 1200 | 600 |
| ZsGreen | 110 | 50 | 350 | 150 |
| DsRed | 200 | 30 | 2000 | 500 |
| Gemini EM | ||||
| AcGFP | 500 | 300 | 1300 | 620 |
| ZsGreen | 110 | 65 | 720 | 170 |
| DsRed | 110 | 30 | 4200 | 200 |
表2. 結果は#cells/wellで表した。cells/mm2に変換するには、96ウェルの値を129 mm2/ウェルで、384の値を25 mm2/ウェルで割る。
一般に、プロットはわずかに非線形で、エンドツーエンドで低下した。これは、高密度のセルがマイクロプレートの壁を遊走し、ビームから外れる傾向があるためではないかと推測される。
表2に、各セルラインの検出下限(LLD)の推定値を示す。LLDの計算は以下の通りである: 3SDBlank/勾配、ここでSDBlankはブランクウェル(細胞を含まない培地)の標準偏差、勾配は下端の曲線の傾きである。(96ウェルプレートと384ウェルプレートでは、それぞれ10,000細胞/ウェルと2500細胞/ウェルのデータポイントを使用した)。非トランスフェクト細胞を封じ込めたウェルのRFUシグナルと標準偏差は、培地のみを封じ込めたウェルと同様であった。
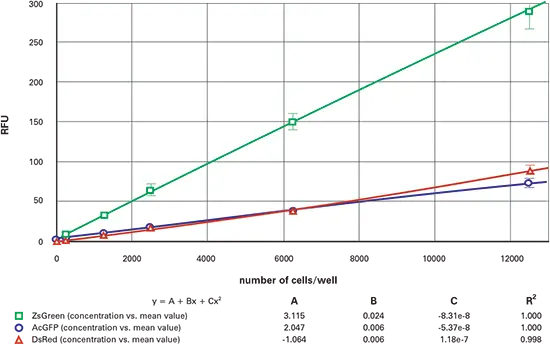
図7. 384ウェル蛍光細胞希釈系列、上から読む。384ウェルプレートにおけるZsGreen(緑)、AcGFP(青)、DsRed(赤)トランスフェクト細胞の希釈系列(上から読む)。
この特定の実験では、ZsGreenとDsRedのセルラインは下方測定を行った場合、同程度の検出限界であり、どちらもAcGFPラインのLLDより3~4倍低かった。この実験は3回実施されたが、DsRedは必ずしも良好な結果を示さなかった。ある実験ではLLDはAcGFPと同程度であったが、別の実験ではLLDがはるかに高かった。このような違いは、細胞株によって継代回数が異なることに起因する。(DsRed細胞株は、初期の実験では他の細胞株よりも成長が早かったため、最初の実験では他の細胞株よりも継代回数が2~4回多かった。
AcGFP細胞株とZsGreen細胞株では、上から読むとLLDが約3倍高かった。一方、DsRedは上から読むと10-20倍高かった。この感度の低下は、蛍光シグナルが著しくレッドシフトするDMEMによる干渉のためと考えられる。実際、着色培地を無色のHBSSに置き換えると、DsRedの上面読み取りの結果は改善した。
表3は、培地を除去して無色の緩衝液に置き換えた以前の実験の結果を示している。下方測定を行った結果には変化がなく(AcGFP LLD は改善されたようであるが)、操作の結果、細胞の著しい損失がなかったことを示している。しかし、トップリードの結果は約3倍改善した。これらの結果は、トップリード・モードにおける感度の低さの一因が着色培地にあるという仮定を支持するものである。
| 下方測定を行う | トップリード | |||
|---|---|---|---|---|
| 蛍光体 | メディア | バッファ | メディア | バッファ |
| AcGFP | 900 | 250 | 16,000 | 3300 |
| ZsGreen | 40 | 70 | 1400 | 400 |
| DsRed | 750 | 600 | 8200 | 3800 |
表3. Gemini EMマイクロプレートリーダーにおける96ウェルマイクロプレートの検出限界(#Cells/Well)に対する培地の無色HBSSへの置換の影響。
セルミックス
この実験では、96ウェル・プレートの各ウェルに様々な混合セルを封じ込め、各ウェル内の細胞総数を50,000個とした。したがって、1:1の混合物には各細胞種25,000個が封じ込められ、1:1:1の混合物には各細胞種16,700個が封じ込められた。プレートはEx/Em 480/510(AcGFPとZsGreenに最適)と550/588(DsRed設定)で読み取られた。観察されたRFU値は予測値と同様であった。この結果は、AcGFP または ZsGreen が DsRed の存在下でも測定でき、その逆も可能であることを示している(表 4)。
| Cells | RFU観察値/予測値(%) |
|---|---|
| AcGFPとZsGreenでの結果: 設定 (480/510) | |
| Ac + Ds | 92.8 |
| Zs + Ds | 99.0 |
| Ac + Ds + Zs | 97.8 |
| DsRedでのRFU結果:セッティング(550/588) | |
| Ac + Ds | 94.9 |
| Zs + Ds | 83.7 |
| Ac + Ds + Zs | 99.6 |
表4. 混合セルで得られた結果。
結論
Molecular Devicesのマイクロプレートリーダーは、接着細胞内の蛍光タンパク質を簡単に検出することができます。それぞれの蛍光色素に対して最適な結果が得られるように波長を調整できるという利点がある。下方測定の方が感度が良いが、上方測定の方が、特にZsGreenとDsRedを含む細胞株で有用な結果が得られる。DsRed細胞株のトップリード結果は、培地を無色のHBSSバッファーに置き換えることで改善できる。
PDF版(英語)