Application Note QuickDrop分光光度計を用いた核酸の定量法および分析
- 最小0.5μLのサンプル使用量
- 1.0ng/μLから2,500ng/μLまでの正確なDNA定量
- スタンドアロンで実験とデータ解析が可能なLCDタッチスクリーン
- キュベット測定用のカイネティックおよびウェーブスキャン機能
PDF版(英語)
はじめに
分光光度法は、生物学的物質の定量や分析に用いられる確立された技術である。その中でも核酸は、ライフサイエンス研究室で最も日常的に測定される物質の一つである。これらのサンプルの濃度と純度を決定することは、PCR、qPCR、シークエンシング、DNAマイクロアレイなどの多くの下流実験にとって極めて重要である。
核酸は主に260 nmで紫外線を吸収し、その濃度は消衰係数とサンプルパス長を関連付けたランベルト・ベールの法則を適用することで計算できる。まず、波長260nmの紫外線を試料に照射し、この光が試料を通過する際に、反対側にある光検出器でどれだけ光が吸収されたかを測定する。その結果を基準(通常はサンプル希釈液)と比較することで、サンプルの核酸の定量法がわかる。
サンプルの純度は核酸定量において重要なパラメーターである。サンプルの純度を決定する最も正確な方法ではありませんが、A260/A280比およびA260/A230比は、タンパク質や化学物質の混入の概算値として使用できます。
SpectraMax® QuickDrop™分光光度計は、核酸サンプルの分析に卓越した多用途の紫外可視(UV-Vis)微量分光光度計です。0.5mmマイクロボリュームポート、キュベットポート、内蔵LCDタッチスクリーンを封じ込め、ユーザーは様々な実験をオール・イン・ワン・システムで行うことができます。
このアプリケーションノートでは、QuickDrop分光光度計が核酸サンプルの定量法(濃度)と定性法(サンプルの純度)の両方を、高い精度と一貫性で分析できることを実証します。
材料と方法
- QuickDrop UV-Vis分光光度計(Molecular Devices社製 cat.)
- UltraPure™ Calf Thymus DNA Solution(Thermo Fisher 社、商品番号 15633019)
- RNA Control 250(Thermo Fisher社のカタログ#AM7155)
- 10 mm 遠紫外線石英キュベット (Starna Cells 社 cat. #9-Q-10)
サンプルのキャリーオーバー
サンプルキャリーオーバーは、子牛胸腺 DNA と超純水でマイクロボリュームポートを交互に測定することで評価した。超純水はリファレンスとして使用した。マイクロボリュームポートは、各測定後にリントフリーワイパーで拭き取りました。
標準曲線の直線性
2500ng/μLの二本鎖DNA(dsDNA)から始まる2倍希釈系列を超純水で調製した。超純水をブランクとして使用し、各サンプル濃度を0.5mmマイクロボリュームポートで3回読み取った。あらかじめプログラムされたDNA定量法を用いて、230 nmから320 nmのWavelengthsにわたって吸光度を測定した。DNA定量メソッドは、以下に示す式に基づいてdsDNA濃度を自動的に算出した。ここで、A260とA320は、それぞれ260 nmと320 nmで測定した吸光度を示す。
μg/mL濃度 = ( A260 -A320 ) x 希釈倍率 x 50 μg/mL
QuickDropは自動的にこれらの計算を行い、ユーザーに濃度を報告する。その後、SoftMax® ProソフトウェアとSoftMax Proインポート機能を使用してデータをグラフ化しました。log-logカーブフィット処理を行い、標準曲線の線形性を示しました。
サンプル量の比較
子牛胸腺DNAは、ストックの子牛胸腺DNAを超純水で希釈して調製した。0.5 μL、1.0 μL、2.0 μLのサンプルをマイクロボリュームポートでテストした(n = 5)。超純水はリファレンスとして使用した。リファレンス容量は、テストしたサンプル容量と同様であった。
RNAバリデーション
ThermoFisher社のRNA Control 250を使用して、QuickDropの分光光度計の性能をNanoDrop™分光光度計の性能と比較しました。キットに付属の保存液をリファレンスとして使用しました。リファレンスサンプルとRNAコントロールの両方をマイクロボリュームポートで測定しました。結果はRNAコントロールキットに付属の仕様と比較しました。
汚染物質の検出
サンプルDNAをフェノール溶液で汚染し、溶液中の最終濃度を50μMフェノールとした。汚染されたサンプルとコントロールは、"Wavescan "機能を使用してキュベットポートで分析されました。この機能は、波長範囲にわたってサンプルの吸光度を段階的にチェックします。結果はMicrosoft® Excelを使用してグラフ化した。
結果
サンプルのキャリーオーバー
サンプルキャリーオーバー実験をQuickDropのマイクロボリュームポートでテストしました。図1と表1の結果から、リントフリーのウェットティッシュで軽く拭くだけで、その後の測定でサンプルのキャリーオーバーが発生しないことが実証されました。
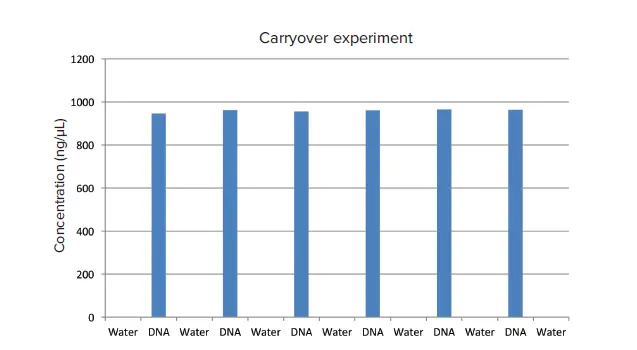
図1. サンプルのキャリーオーバー。マイクロボリュームポートで子牛胸腺DNAと水を交互に測定。データはExcelでグラフ化した。このグラフを構成するデータを表1に示す。
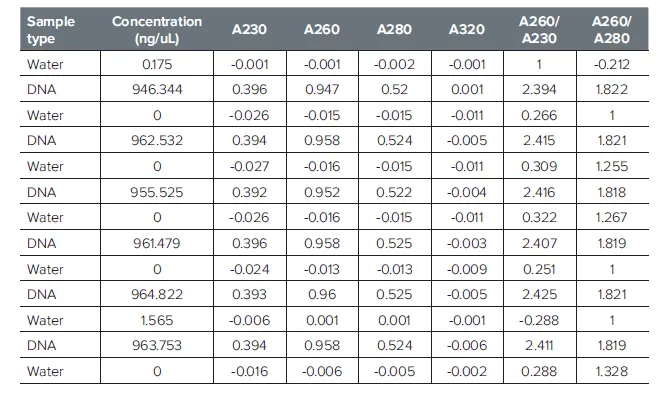
表1: 図 1 の「サンプルキャリーオーバー」実験を構成するデータ。
標準曲線の直線性
図2において、QuickDropはDNA濃度と希釈倍率の間に直線関係を示しました。この実験から、1 ng/μL DNAがこのQuickDropで測定可能な最低濃度であると判断しました。
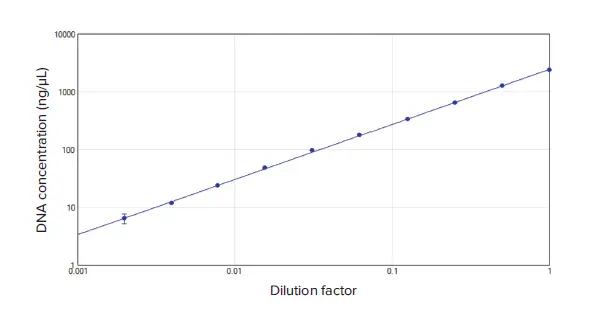
図2. 計算されたDNA濃度と希釈倍率の関係。2500ng/μLから始まる子牛胸腺DNAの2倍希釈系列は、希釈倍率とQuickDropによって報告されたDNA濃度との関係を示している。曲線は優れた直線性を示し、r2値は1.000であった。
サンプル量の比較
3種類の異なる容量のサンプルDNAをQuickDropのマイクロボリュームポートでアッセイしました。容量の違いにもかかわらず、算出されたサンプルDNA濃度は一貫していました(図3)。最良の結果を得るためには、ピペッティングが最も簡単であった2μL容量の使用をお勧めします。
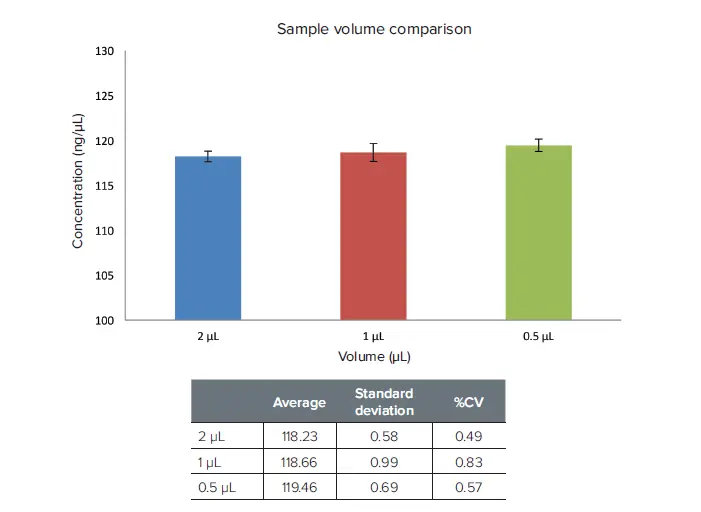
図3. 容量の再現性。2 μL、1 μL、0.5 μL容量の子牛胸腺DNAをマイクロボリュームポートで読み取った。異なる容量でも、計算濃度は非常に類似していた(n = 5)。標準偏差と%CVは、すべてのサンプル容量で非常に類似していた。
RNAバリデーション
QuickDropのRNA測定能力を実証するため、RNA 250コントロールを測定しました。測定された濃度は、コントロールキットが提供する許容範囲内でした(250±5ng/μL)。
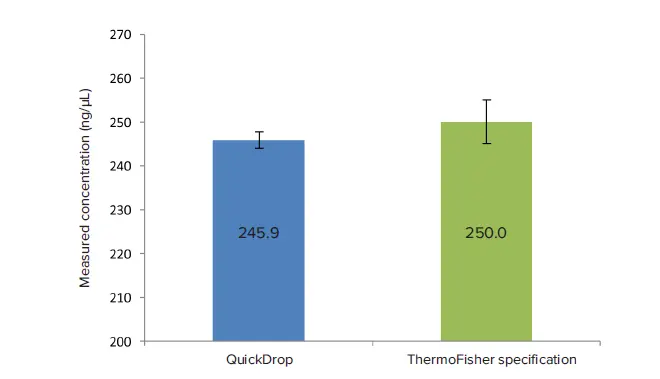
図4. RNAバリデーション実験。250ng/μLのRNAコントロールをQuickDropで測定し、ThermoFisherのNanoDropの検証結果と比較した(n = 4)。測定されたRNA濃度(245.9 ± 1.9 ng/μL)はThermoFisherの規定範囲(250.0 ± 5.0 ng/μL)内であった。
汚染物質の検出
Wavescan機能を使用すると、コントロールと比較して230 nmの吸光度が増加するため、サンプル中の汚染物質を特定することができます(図5)。この機能は、グアニジンチオシアネートなど、DNA精製プロセスで検出される他のタイプの汚染物質にも適用できます。QuickDropの有効波長範囲は190 nmから1100 nmまであり、包括的な化学分析が可能です。
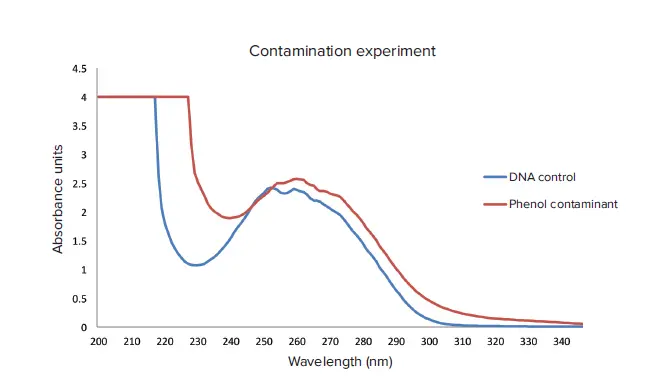
図5. 汚染物質実験。フェノールで汚染された試料と汚染されていない対照試料をWavescan機能で測定。Microsoft Excelを使用して、Wavescanのトレースをグラフ化し、重ね合わせた。230 nmの吸光度に明確な増加が見られる。
結論
QuickDropの内蔵キュベットポートとマイクロボリュームサンプルポートは、核酸サンプルの高感度定量法と分析法の両方を可能にします。上に示したように、わずか0.5μLのサンプルでもマイクロボリュームポートで安定して読み取ることができます。また、検出範囲が広く(1ng/μL~2500ng/μL)、サンプルキャリーオーバーが少ない。
最後に、内蔵のLCDタッチスクリーンは、サンプルの濃度や純度などの重要な情報を研究者に提供する(図6)。これらすべての特徴と設置面積の小ささにより、QuickDrop分光光度計は、どのようなラボ環境においても核酸の定量法と分析に最適な選択肢となります。

図6. QuickDropのデータ表示例。関連するパラメータが計算され、QuickDropのタッチスクリーンに表示される。
SpectraMax QuickDrop微量分光光度計について詳しく>>
PDF版(英語)