Application Note 合成メタゲノミクス:デジタル情報を生物学に戻す
- コロニーの検出と蛍光マーカーの定量を同時に実施 - 表現型セレクションを使用してコロニーをピッキング
- プレーティング、ストリーキングからピッキングまでの自動ワークフロー ・サンプルの散布からピッキング、複製、再アレイ化までのサンプル履歴を記録・追跡
- サンプル散布からピッキング、複製、再アレイ化までのサンプル履歴を記録・追跡。
- 1日あたり最大30,000コロニーを98%以上の効率で正確にピッキング
PDF版(英語)
はじめに
米エネルギー省合同ゲノム研究所(DOE JGI)の研究者たちは、次世代シーケンサーが生み出す膨大な「デジタル」データを、具体的な観察可能生物学に変換するために、ハイスループットスクリーニング技術に支えられた合成メタゲノム研究プロセスを開発した。
次世代シーケンサー技術により、既知の経路や機能活性のフルスペクトルを網羅する数百万もの新規予測遺伝子が同定された。これらには、エネルギーや環境技術への応用が期待される新規の生体触媒や、基礎研究や応用バイオテクノロジーにおける潜在的な用途が多数含まれている。主要な課題は、膨大な量の「デジタル」情報(配列決定、生物製剤、化学情報)を、観察可能な機能生物学(酵素活性、細胞死、タンパク質発現など)に変換することである。
DoE JGIは、この課題を克服するために合成メタゲノミクス・プロセスを開発した。遺伝子とパスウェイは、テンプレートに依存しない方法で合成され、それによってデジタル情報を生化学的分子に、迅速でコスト効率の良い方法で変換する。
セルロース系バイオ燃料の商業的生産の大きなボトルネックは、バイオマスからグルコースへの変換に十分な活性とロバスト性を持つ酵素がないことである。イオン液体は、セルロースを溶解する前処理として使用することができ、グルコースのような発酵可能な糖の利用可能性を高める。工業的に適切な酵素候補を見つけるために、合成メタゲノムアプローチが用いられた。DoE Joint Bioenergy Institute (JBEI)で設計された特異性産業応用のためには、(i)70℃での機能的酵素活性、(ii)pH4.5での安定性、(iii)イオン液体に対する耐性、という仕様を持つ酵素が必要であった。プロジェクト開始時点では、必要な条件下で十分な活性を示す酵素は知られていなかった。
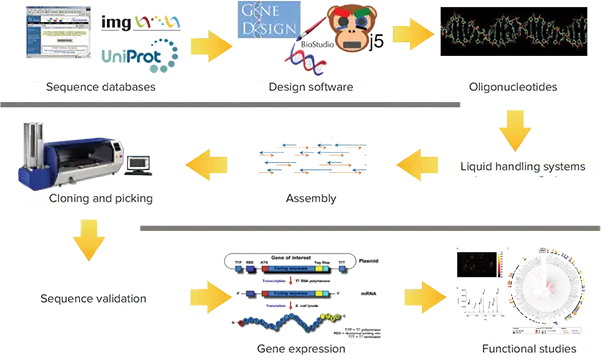
図1. 合成メタゲノミクスパイプライン:DoE JGIがメタゲノム研究に適応したプロセス。メタゲノム研究における典型的な合成スキーマ。クローニングとピッキングの段階でQPix 460システムを使用することで、ワークフローのタイムラインが大幅に短縮され、労働集約的でエラーの起こりやすい手作業を自動化することで効率が向上する。完全自動ワークフローは、プレーティングからピッキングまで、週に数千のコロニーを正確に処理することができ、完全なデータ追跡により、塩基対あたりのコストを削減します。
最初の研究対象は、セロビオースからグルコースへの変換を仲介するGH1として知られる酵素ファミリーであった。系統的誘導アプローチにより、利用可能な数千の配列から、機能的多様性が最大となる200遺伝子を選択することができた。200遺伝子のうち、180遺伝子を合成し、QPix 460システムを用いて発現ベクターにクローニングした(図2)。
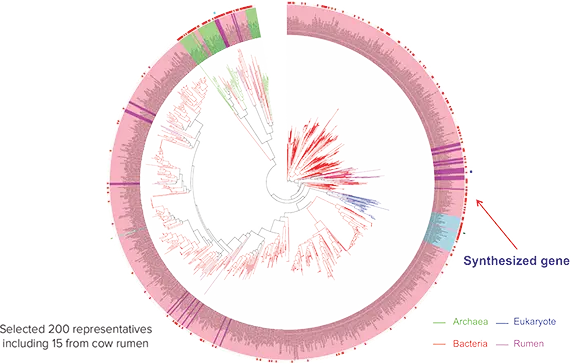
図2. 代表的な遺伝子ファミリーを選択するためのメタゲノム系統解析。細菌(赤)、真核生物(青)、牛のルーメン(紫)、古細菌/「極限原核生物」(緑)を含むメタゲノム樹形図。枝の長さが長いほど遺伝的多様化が進むが、短い場合はそうではない。分岐の短い領域から200の代表的なサンプルを選択し、その中には系統学に基づくサンプルの多様性に貢献するため、牛のルーメンから15を含む。
DoE JGIで開発されたハイスループットの自動ワークフローでは、形質転換、プレーティングを並行して行い、配列検証のために各遺伝子変異体から複数のクローンをピッキングする。これは、ハイスループットのプレーティング、ストリーキング、コロニーピッキングを単一プラットフォームで提供するQPix 460システムによって可能となった。配列が確認されると、各遺伝子について所望のクローンがタンパク質生産株に移植された。
クローニングされたGH1タンパク質生産株を可溶性タンパク質発現について分析した。クローンの65%が可溶性タンパク質を産生し、このタンパク質を上記の必要な条件下でさらに特性評価した。例えば、初期の品質制御として、多くの酵素を異なる温度での活性について特性評価し、耐熱性を持つ候補を明らかにした(図3)。
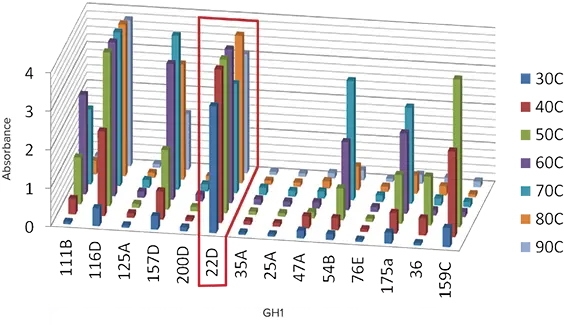
図3. ファミリーGH1酵素活性の解析から、耐熱性酵素が明らかになった。比色分析基質に対する酵素活性を光学密度で測定。タンパク質は30℃から90℃までの7つの異なる温度で酵素活性をアッセイした。最適な候補(例えば22D)は、70℃では活性がほとんどあるいは全く低下しない。
すべての選択基準を評価するのに必要な1万5千もの反応をスクリーニングするには、超ハイスループット技術が必要であった。この目的のために、質量分析1 に基づくハイスループット技術であるナノ構造イニシエーター質量分析法(NIMS)が用いられた。
酵素-基質反応を固体支持体上にスポットし、レーザーでイオン化した。
酵素活性は、基質と生成物の比率を定量することによって決定された(図4)。この大規模なデータセットから、詳細な特性解析のために、活性の高い上位20種の酵素が選ばれ、そのうちの5種は、上述の工業的に適切な条件下で活性を示した。このことは、合成メタゲノミクスを用いることで、公開配列データベースの予測タンパク質から出発して、ユニークな特性を持つ酵素を迅速に同定できることを示している。
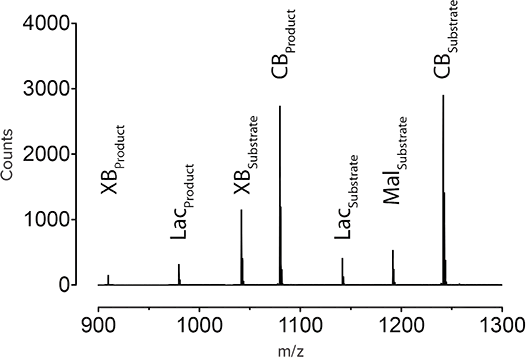
図4. NIMSに基づく基質:生成物の比率。NIMS分析により、基質と生成物の比率で測定した基質との最適な反応性に基づいて、酵素の性能上位20種が明らかになった。
結論
この研究は、DoE JGIが開発し利用したプロセスにより、デジタル情報を生物学的に関連性のある、新しい特性を示す機能的酵素に変換することに成功したことを示している。GH1 酵素ファミリーの 180 種多様なタンパク質が、QPix 460 システムを利用したハイスループット自動化アプローチでスクリーニングされた。このシステムは、効率的なメタゲノムライブラリーの作製と管理のための自動ワークフローに不可欠な部分となった。
さらに、このプロセスにより、70℃、pH4.5、イオン液体存在下で、工業的に関連性の高い基質に対して活性を示す5つの新規酵素候補が同定された。ジョイント・バイオエナジー研究所(JBEI)は現在、バイオマスをグルコースに変換する生産を効果的にスケールアップするため、これらの酵素を産業環境に応用する可能性を研究している。
参考文献
-
ナノ構造-イニシエーター質量分析に基づく酵素活性アッセイ。Northen TR, Lee JC, Hoang L, Raymond J, Hwang DR, Yannone SM, Wong CH, Siuzdak G. Proc Natl Acad Sci USA. 2008 Mar 11;105(10):3678-83. doi: 10.1073/pnas.0712332105. Epub 2008 Mar 4. PMID:18319341 [PubMed - Indexed for MEDLINE].
PDF版(英語)