Application Note Gemini XS、Gemini EM、SpectraMax M2マイクロプレートリーダーで
OliGreenオリゴヌクレオチド定量試薬を使用する場合
PDF版(英語)
はじめに
Molecular Devices Corporation, 1311 Orleans Dr., Sunnyvale, CA 94089のYan Zhang氏によるもの。

このアプリケーションノートでは、Molecular Devices社のSoftMax® Proソフトウェアを搭載したGemini XS、Gemini EM、SpectraMax® M2マイクロプレートリーダーで、Molecular Probes社のOliGreen® 定量キットを使用する方法について説明します。一本鎖 DNA とオリゴヌクレオチドは、従来 260 nm で吸光度を測定していました。OliGreen は、従来の光度計を用いた方法よりもはるかに高感度で特異性があります。マイクロプレートフォーマットでのこのアッセイのダイナミックレンジは、Gemini XSで読み取れる0.05 ng/mlから1 µg/mlです。このアプリケーションノートに記載されたデータは、モレキュラー・プローブ社のアプリケーションノート(MP 07582)に記載されたダイナミックレンジと検出下限を裏付けるものです。
OliGreen のスペクトルは、Molecular Probes 社の製品添付文書に記載されているように、励起/Emission: 500/520 nm です。しかしながら、励起/ Emission 波長は 480/520 nm を使用することを推奨しています。Gemini XSのデュアルモノクロメーターは1 nm単位で任意の波長を選択できるため、シグナルを最適なダイナミックレンジで得るために、複数の異なる励起/発光波長でアッセイプレートを読み取った。Gemini XSで使用された最適な励起/発光波長: 480/520、515 nmでカットオフ。
材料
Molecular Probes社製 OliGreenオリゴヌクレオチド定量試薬 (Cat.# O-7582)
オリゴヌクレオチド標準:Molecular Probes社製M13シーケンスプライマー(Cat.# O-11492)
黒の96ウェルプレート
茶色のエッペンドルフチューブ
分光蛍光光度計(Gemini XS、Gemini EMまたはSpectraMax M2)
SoftMax Proソフトウェア(バージョン4.5以降)
方法
インストゥルメンテーションとソフトウェアのセットアップ
ステップ1:分光蛍光光度計の電源を入れます
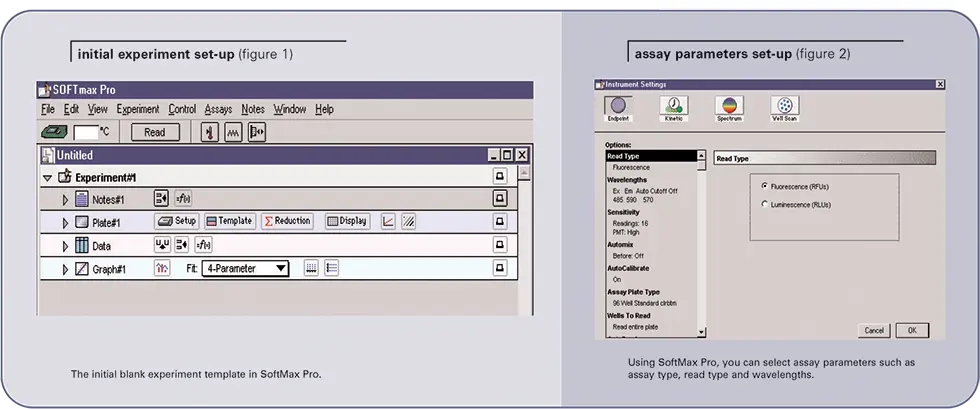
ステップ2: SoftMax Proソフトウェアを起動し、新規ファイルを開きます。(図1を参照)。
ステップ3:"Set Up "をクリックします。二つ目の画面が表示されます。(図2参照)まず、読み取りタイプを蛍光に選択し、画面左側の各オプションでアッセイに適したパラメータをすべて設定します:波長(EX 480、EM 520、カットオフ515)、感度30(数値が高いほど高精度が得られますが、プレートの読み取りに時間がかかります)、アッセイプレートタイプ、読み取りウェルなど。
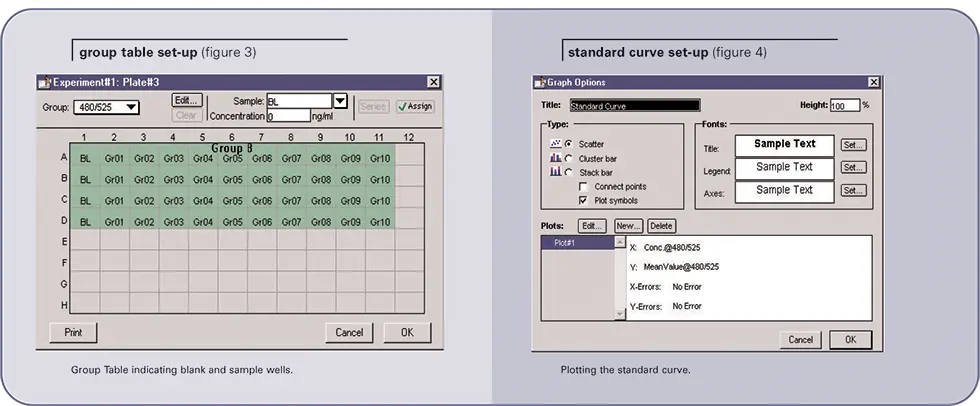
ステップ4:マイクロプレート上のブランクウェルとサンプルウェルの位置を示すアッセイのテンプレートを作成します。(図3参照)。
アッセイの準備
このアッセイの方法は、Molecular Probes 社の OliGreen ssDNA 定量試薬の製品情報シートに記載されている指示 に従いますが、96 ウェルマイクロプレートフォーマットに適合させるため、アッセイ容量を 2.0 ml から 200 µl に減らすことを除きます。
ステップ1:キットの濃縮バッファー20倍を蒸留DNaseフリー水で希釈し、1x TE(10 mM Tris-HCl、1 mM EDTA、pH 7.5)バッファーを調製する。
ステップ2: 100μg/mlのストック(キットから)から2μg/mlと20ng/mlのオリゴヌクレオチド溶液を1x TEバッファーに調製する。
ステップ3:マイクロプレート上のブランクウェルとサンプルウェルの位置を示すアッセイのテンプレートを作成する。(図3参照)。
ステップ4: 2μg/mlと20ng/mlのストック溶液を希釈します。1x TEバッファーで表1のように連続希釈する。プレートのブランクとして使用する0 ng/ml濃度の試料を含む。プレートブランクのRFU値は、プレートの全サンプルのRFUから差し引く。
ステップ5: 濃縮ストックを 1X TEバッファーで 1:200 に希釈し、実験の直前に OliGreen 試薬の作業溶液を調製する。Molecular Probesの推奨に従い、アルミホイルで覆って光から保護する。
ステップ6:適切なオリゴヌクレオチド溶液とOliGreen作業溶液を、以下の表に記載されているように、11本の茶色(遮光用)のエッペンドルフチューブに混ぜる:
| 表1. オリゴヌクレオチド希釈 | |||
|---|---|---|---|
| オリゴヌクレオチド濃度(ng/ml)) | オリゴヌクレオチド量 | 1x TEバッファー溶液(µl) | 最終アッセイ オリゴヌクレオチド濃度 (ng/ml) |
| 0(空白) | 0 | 1000 | 0(空白) |
| 0.1 | 5 µl of 20 ng/ml | 995 | 0.05 |
| 0.4 | 20 µl of 20 ng/m | 980 | 0.2 |
| 1 | 50 µl of 20 ng/ml | 950 | 0.5 |
| 2 | 100 µl of 20 ng/ml | 900 | 1 |
| 4 | 200 µl of 20 ng/ml | 800 | 2 |
| 20 | 10 µl of 2 ng/ml | 990 | 10 |
| 40 | 20 µl of 2 ng/ml | 980 | 20 |
| 200 | 100 µl of 2 ng/ml | 900 | 100 |
| 400 | 200 µl of 2 ng/ml | 800 | 200 |
| 2000 | 1000 µl of 2 ng/ml | 0 | 1000 |
ステップ6:各濃度のオリゴヌクレオチド100μLをマイクロプレートのウェルにトリプリケートで加える。次にOliGreen試薬のワーキングストックを100μLずつマイクロプレートの各サンプルウェルに加える。
注意:OliGreen 試薬を添加した後は、常にプレートを遮光してください。
マイクロプレートを読む
ステップ1:マイクロプレートをリーダーにセットする。
ステップ2:測定値を読み取る前に、プレートをインストゥルメンテーションで5分間インキュベートします。
ステップ3:ソフトウェアのReadボタンをクリックします。スペクトロフルオロメーターがプレートを読み取り、相対蛍光単位がプレートセクションに表示されます。
データを分析する
ステップ 1: マイクロプレートの読み取りが終わると、RFU が Plate セクションに表示されます。データは、テンプレートのセットアップ中に作成したグループ表で分析されます(2ページの「インストゥルメンテーションとソフトウェアのセットアップ」のステップ5を参照)。グループテーブルの例については、本書の結果セクションを参照してください。
ステップ2:サンプルの平均RFUをオリゴマー濃度に対してプロットします。(図4参照)。
ステップ3:グラフセクションのツールバーにあるドロップダウンカーブフィットメニューから適切なカーブフィット処理を選択します。標準曲線をアッセイのダイナミックレンジ全体にプロットする場合はlog-logカーブフィットを使用します。標準曲線のごく一部をプロットする場合は、線形カーブフィットを使用します。
波長の最適化
プレートをいくつかの異なるExcitation/Emission波長の組み合わせで読み取り、データを解析した。(図7参照)。
結果
Molecular ProbesのOliGreen試薬とランダム配列35-Merオリゴヌクレオチドを用いて、以下の標準曲線を得た(図5参照)。(図5参照)。
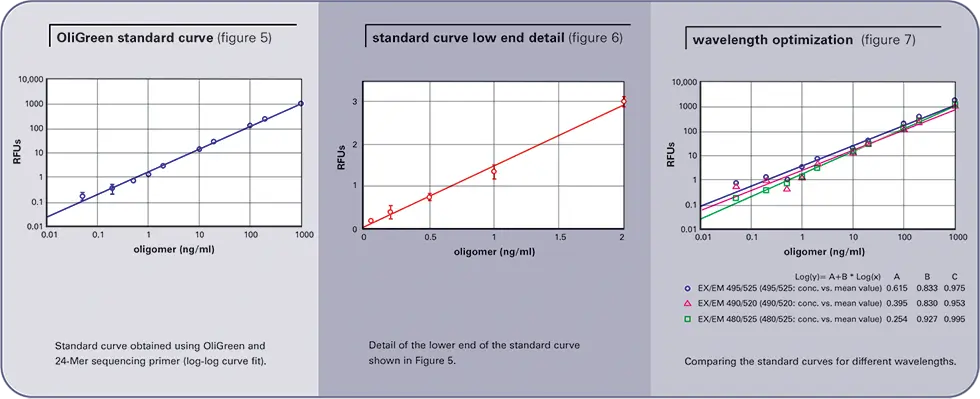
Molecular Probes社では、蛍光マイクロプレートリーダーで測定した場合の検出下限を1 ng/mlとしている。我々が収集したデータは、この検出下限を裏付けるものである。標準曲線の下限を図6に示す。
495/525または490/520で励起/Emissionを使用することは、ノイズと信号のダイナミックレンジの点で、480/525ほど良くない。(図7参照)Gemini EMとSpectraMax M2から同一のデータと曲線が生成されたが、ここでは示していない。
結論
Molecular ProbesのOliGreenアッセイは、SoftMax Proソフトウェアを搭載したGemini XSまたはEM蛍光マイクロプレートリーダーで実行すると、一本鎖DNAの迅速で高感度な検出メソッドとなります。SoftMax Proの分析機能により、読みやすく、ユーザーがカスタマイズ可能なレポート形式で定量を行うことができます。Gemini XS、Gemini EM、SpectraMax M2マイクロプレートリーダーは、波長を最適化する柔軟性を備え、最適なリードアウトを保証します。
参考文献
モレキュラー・プローブ データシート MP 7582 02/08/96: OliGreenオリゴヌクレオチド定量試薬(O-7582)一本鎖DNA用。
PDF版(英語)