Application Note マイクロプレート上でのイメージングと
ウェスタンブロット検出を用いたCRISPR編集細胞の検証
- GFP陽性細胞と非導入細胞を正確に識別することによりトランスフェクション効率を測定する。
- タンパク質バンドのウェスタンブロット分析を用いてCRISPRを介した遺伝子ノックダウンを検証する。
- 高品質のウェスタンブロット画像を取得
PDF版(英語)
はじめに
ゲノム編集は、遺伝子発現やタンパク質機能の研究に広く用いられているが、これらの方法の多くは手間がかかり、精度も低い1 。CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9システムは、その精度の高さと使いやすさから、遺伝子編集のためのツールとして非常に普及している2。Cas9システムは、細菌や古細菌の自然免疫システムの一部として誕生し、外来DNAから身を守るために用いられてきた3,4。既知の外来DNAの短い配列は、CRISPRターゲティングRNA(crRNA)として発現され、Cas9酵素が同様の配列を含む外来DNAを切断するようガイドする。このシステムを利用することで、研究者は遺伝子サイレンシングと転写制御の効果を研究することができ、疾患細胞における遺伝的障害を緩和できる可能性がある5。
crRNAに似たガイドRNA(gRNA)は、遺伝子内のある領域を標的として設計され、Cas9酵素は宿主細胞のゲノムのこの特異性領域に二本鎖切断を作り出すことができる(図1)。二本鎖切断が生じると、細胞は非相同末端結合(NHEJ)経路または相同組換え(HDR)経路という2つの修復経路のいずれかを経る。NHEJ経路は一般に、塩基の挿入や欠失(indel)によって遺伝子を破壊するのに使われ、一方、HDR経路は、2つの類似または同一分子のDNA間で配列を交換することによって、レポーター遺伝子や編集配列をノックインするのに使われる6。
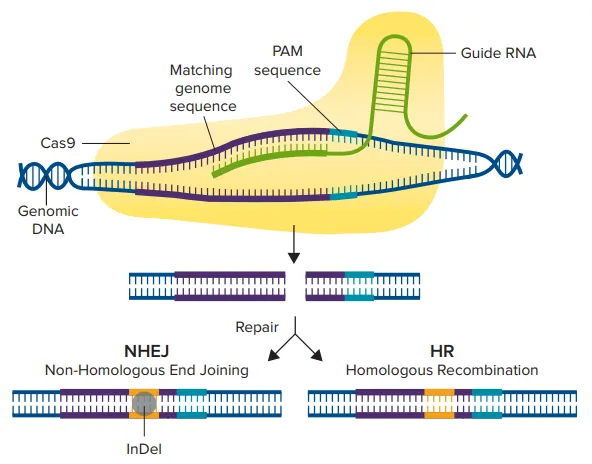
図1. CRISPR/Cas9のメカニズム。Cas9酵素は、まずガイドRNAに結合し、次に3塩基PAM配列の直前にある一致するゲノム配列に結合することで活性化される。その後、Cas9酵素が二本鎖切断を作り、NHEJまたはHDR経路のいずれかがDNAの修復に使用され、編集された遺伝子配列が得られる。
遺伝子編集のバリデーションは、導入された変異が目的の遺伝子をノックダウンしたかどうかを判定するために必要である。通常、欠失や挿入の成功を確認するためにゲノムPCRや遺伝子シーケンシング検査が行われるが、インフレーム欠失/変異からのシグナルを避けるためには、タンパク質発現を測定することが遺伝子ノックダウンのより明確な指標となる7。
ここでは、SpectraMax i3x マルチモードマイクロプレートリーダーとScanL™ウエスタンブロット検出システムを用いて、CRISPR/Cas9ゲノム編集を検証する方法を示します。CRISPRキットを介したOrigeneのATG5ヒト遺伝子ノックアウトキットを用いて、HEK293セルにおけるオートファジー関連5(ATG5)タンパク質の発現をノックダウンし、緑色蛍光タンパク質(GFP)とピューロマイシン耐性遺伝子をノックインした。ノックダウンはScanLaterウェスタンブロット解析で確認した。
材料
- ATG5 Human CRISPR Knockout kit via CRISPR (Origene cat. #KN210563)
- FUGENE HDトランスフェクション試薬 (Promega cat. #E2311)
- Opti-MEM 還元血清培地 (ThermoFisher cat. #31985062)
- ATG5 に対するウサギポリクローナル抗体 (Origene cat. #TA301481)
- ATG5 HEK293T細胞一過性過剰発現溶解液 (Origene cat. #LC401532)
- pCas-Guide-EF1a-GFP プラスミド (Origene cat. #GE100018)
- HEK293細胞(ATCC cat.)
- SpectraMax® i3x Multi-Mode Microplate Reader (Molecular Devices cat. #i3x)
◦ScanLater™ウェスタンブロット検出システム(Molecular Devices社製 cat.)
- SpectraMax® MiniMax™ 300イメージングサイトメーター(Molecular Devices cat.)
- ScanLater: Eu-ストレプトアビジン評価キット(Molecular Devices cat.)
- ScanLater: 抗マウス評価キット(Molecular Devices cat.)
- Pierce BCAタンパク質アッセイキット (Thermofisher cat. #23225)
- ピューロマイシン(InvivoGen cat.)
- TGX 12% SDS-PAGEゲル (Bio-Rad cat. #1620174)
- TransBlot Turbo Mini-size LF PVDFメンブレン (Bio-Rad cat. #1620174)
- Trans-Blot® Turbo™システム (Bio-Rad cat. #1704150)
方法
細胞トランスフェクション
HEK293細胞をCostar組織培養処理6-ウェルプレートに1ウェル当たり30万細胞でプレーティングした。セルは(1)ガイドベクター(AAGATGTGCTTCGAGATGTG)と(2)ピューロマイシン耐性とGFPの配列を含むドナーベクターでトランスフェクトした。並行して、3ウェルの細胞を、トランスフェクションコントロールとしてGFPを一過性に発現するpCas-Guide-EF1a-GFPベクターでトランスフェクトした。6μLのFUGENE HDトランスフェクション試薬と2μgの全DNA(3:1の比率)を用いて細胞をトランスフェクトした。その後、細胞を37℃で一晩インキュベートした。トランスフェクトされたセルはGFPを発現し、MiniMaxサイトメーターの緑色蛍光チャンネルで同定・計数された。総細胞数はStainFree解析を用いて算出し、トランスフェクション効率はSoftMax Proソフトウェアを用いてGFP陽性細胞数を細胞総数で割ることにより測定した。
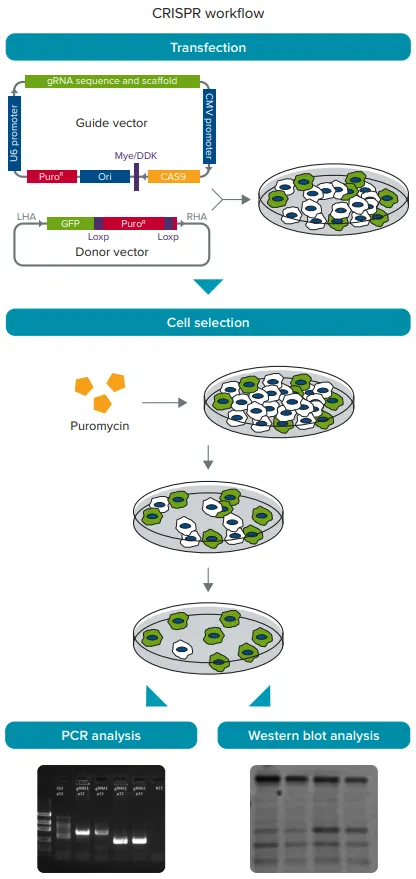
図2. CRISPR/Cas9実験ワークフロー。HEK293細胞にガイドベクターとドナーベクターをトランスフェクト。次に、遺伝子が挿入された細胞を濃縮するために、細胞を1.0μg/mLのピューロマイシンで選択する。選択したセルからDNAとタンパク質を回収し、ゲノムPCRとウエスタンブロッティングを行い、それぞれ遺伝子の挿入とノックダウンを確認する。
セル選択
トランスフェクトした細胞を1.0 µg/mLのピューロマイシンを含む培地で培養し、挿入されたピューロマイシン耐性遺伝子を持つ細胞を選択した。その後、セルを溶解し、ゲノムDNAと総タンパク質を回収した。ゲノムDNAはPCR解析のためにGENEWIZに送られ、遺伝子が正しく挿入されていることを確認した。タンパク質濃度は、SpectraMax i3xリーダーで読み取ったPierce BCAアッセイを用いて定量し、結果はSoftMax Proソフトウェアで事前に設定したプロトコルを用いて解析した。
ウェスタンブロット
細胞溶解液をLaemmliサンプル緩衝液で希釈し、100℃で10分間煮沸することにより、CRISPR編集細胞と非編集細胞の両方から5μgと10μgの全タンパク質を調製した。サンプルを4-20%プレキャストゲルにローディングし、SDSPAGEを行った。その後、Trans-Blot Turboシステムのセミドライ法を用いてタンパク質をPVDFメンブレンに転写した。
メンブレンはScanLaterブロッキング緩衝液中、室温で1時間インキュベートした。その後、メンブレンのローディング対照、ラダー、ATG5領域のレーンを別々に切り分け、それぞれの一次抗体(表1参照)中で4℃、一晩インキュベートした。ScanLater洗浄緩衝液で3回洗浄した後、ブロットをScanLater Eu標識二次抗体と室温で1時間インキュベートした。抗体とその希釈倍率を表1に示す。
| 一次抗体 | 二次抗体 | |
|---|---|---|
| サンプルブロット | 1:2500 rabbit anti-ATG5 | 1:10000 Goat Anti-Rabbit |
| ローディング対照 | 1:10000 rabbit anti-Vinculin | 1:10000 Goat Anti-Rabbit |
| ScanLaterウェスタンブロットタンパク質ラダー | 1:10000 Eu-Streptavidin |
表1. ウェスタンブロッティングに使用した抗体の希釈率。
二次抗体インキュベーション後、メンブレンをScanLater洗浄緩衝液で3回洗浄し、乾燥させ、ScanLater™ウェスタンブロット検出システムで読み取る前に再度組み立てた。
データ分析
データはSoftMax® ProソフトウェアからImageJソフトウェアにエクスポートされ、相対的なATG5タンパク質発現を定量するためにバンド密度が測定された。細胞-細胞および細胞-マトリックス接合部に関連する117kDaの細胞骨格タンパク質であるビンキュリンを、サンプルローディングのばらつきを正規化するためのローディング対照として使用した。
結果
MiniMax細胞計はトランスフェクトした細胞とトランスフェクトしていない細胞の両方の高画質画像を撮影し、SoftMax® ProソフトウェアはGFP陽性細胞の数と細胞の総数を同定し、定量することができた。トランスフェクション効率は13%と算出された(図3)。
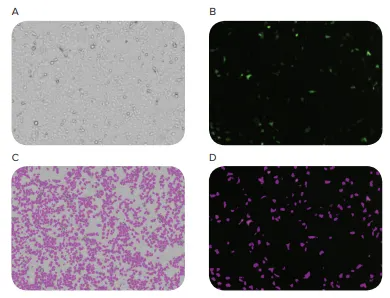
図3. 細胞トランスフェクションの画像。HEK293細胞をpCas-Guide-EF1a-GFPベクターでトランスフェクトし、CRISPR/Cas9トランスフェクト細胞と並行してトランスフェクション効率を計算した。MiniMaxサイトメーターを用いて、明視野チャンネル(A)と緑色蛍光チャンネル(B)で細胞をイメージングした。SoftMax Proソフトウェアで両チャンネルの細胞を同定・計数し(CとD)、GFP陽性細胞を全細胞数で割って13%のトランスフェクション効率を算出した。
ゲノムDNAサンプルのPCR解析から、HEK293細胞ゲノムのATG5領域に正しいサイズの挿入がなされたことが判明した(データは示さず)。
ウェスタンブロット画像では、CRISPR編集細胞と非編集細胞(陰性対照)との間で、ATG5タンパク質バンド強度の目に見える減少が見られた(図4)。ImageJソフトウェアを用いてタンパク質のバンド密度を計算すると、CRISPR編集細胞は未編集細胞と比較して、ATG5タンパク質の発現がおよそ60%ノックダウンしていた(図5)。
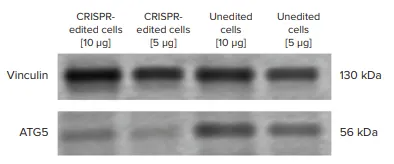
図4. ATG5のウェスタンブロット実験。ATG5タンパク質の発現は、編集していないセルに比べてCRISPRしたセルでは目に見えて低かった。サンプルローディングの過不足を考慮し、5μgと10μgのサンプルをSDS-PAGEゲルにロードした。サンプルローディングのばらつきを正規化するため、ローディング対照としてVinculinを用いた。ウェスタンブロットの画像をImageJソフトウェアにエクスポートし、バンド密度を測定した。
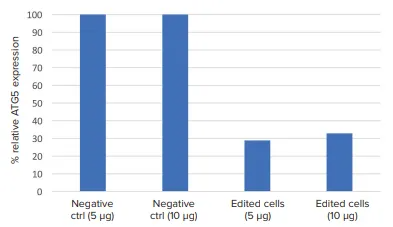
図5. 相対的ATG5タンパク質発現。図4のウエスタンブロット画像をImageJソフトウェアにエクスポートし、バンド密度を解析した。ATG5タンパク質のバンド密度は、それぞれのローディング対照に対して正規化した。CRISPR編集細胞は、非編集細胞と比較してATG5タンパク質発現が一貫して低かった。
結論
CRISPR遺伝子編集技術では、正確な結果を得るためにプロセス全体を注意深くモニタリングする必要があります。SpectraMax i3x マルチモードマイクロプレートリーダーは、最初のトランスフェクションからタンパク質ノックダウンの確認まで、CRISPR編集実験の結果を分析するための完全なソリューションを提供します。MiniMax細胞計を使用すると、研究者は、総非標識細胞数と蛍光発現トランスフェクト細胞数を比較することにより、トランスフェクション効率を評価することができます。ScanLaterウエスタンブロット検出システムは、コントロール細胞とCRISPR導入細胞における目的のタンパク質の高感度検出と定量分析を可能にします。
参考文献
- Lombardo, A et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nature Biotechnology 25.11 (2007): 1298.
- Barrangou, R. Cas9 targeting and the CRISPR revolution. Science 344.6185 (2014): 707-708.
- Ishino, Y et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology 169.12 (1987): 5429-5433.
- Barrangou, R et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science 315.5819 (2007): 1709-1712.
- Barrangou, R et al. Applications of CRISPR technologies in research and beyond. Nature Biotechnology 34.9 (2016): 933-941.
- Liang, F et al. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proceedings of the National Academy of Sciences 95.9 (1998): 5172-5177.
- Estep, JA et al. Immunoblot screening of CRISPR/Cas9-mediated gene knockouts without selection. BMC molecular biology 17.1 (2016): 9.
PDF版(英語)