Application Note SpectraMaxマイクロプレートリーダーでの
KASPジェノタイピングテクノロジーによるSNP検出
- 蛍光マイクロプレートリーダープラットフォームでSNPジェノタイピングが可能
- SNPジェノタイピングのための迅速、コスト効率的、ハイスループットな手法
- SoftMax® Proソフトウェアで多波長蛍光測定値の取得と解析が容易
PDF版(英語)
Young Mee Yoon|フィールドアプリケーションサイエンティスト|モレキュラーデバイス
はじめに
ジェノタイピングとは、DNA配列を調べることにより、個体間の遺伝的差異を分析するプロセスです。ジェノタイピングには多くの方法があり、研究者はヒト、微生物、植物におけるゲノムの多様性を調査することができます。一塩基多型(SNP)は遺伝的多様性の最も一般的なタイプの一つであり、特異性遺伝子座における一塩基の変異からなります。SNPジェノタイピングは、様々な生物種における疾患関連変異の同定に非常に有用であることが証明されており、その結果、SNP検出のための多くの技術が開発されてきました1。
Kompetitive Allele Specific PCR (KASP)はSNPジェノタイピングに最も広く用いられている手法の一つであり、その精度の高さ、コスト効率の良さ、アッセイデザインの柔軟性の高さが評価されています。KASPは2つの対立遺伝子特異的フォワードプライマーと1つの共通リバースプライマーを用いて、ポリメラーゼ連鎖反応(PCR)により標的DNA配列を増幅し、反応産物をその配列に対応する蛍光色素で標識します。サンプルがホモ接合体の場合、PCR産物は蛍光色素HEXまたはFAMのみで標識されるが、ヘテロ接合体の場合はHEXとFAMの両方で標識された産物が存在します。取り込まれなかった蛍光色素はクエンチされる2, 3 。KASPアッセイはマイクロプレートフォーマットで行われ、結果は蛍光マイクロプレートリーダーを用いて検出することができます。
このアプリケーションノートでは、LGC Genomics社のKASPアッセイ検証キットを使用して、SpectraMaxマイクロプレートリーダーをどのようにKASP最終産物の読み取りに使用できるかを示します。
最初の実験では、リーダーがFAM、HEX、FAM/HEXを含む各フルオロフォアに特徴的なシグナルを発生することを検証しました。2回目の実験では、ベンダーから提供された既知のDNAサンプルを用いてKASPアッセイを行い、PCR産物をリーダーで検出し、サンプルのジェノタイピングが正しいことを確認しました。
材料
- 標準 ROX バリデーションキット(LGC Genomics)
- サーマルサイクラー (MJ Research)
- TempPlateセミスカート96ウェルPCRプレート (USA Scientific)
- 384-well ソリッドブラックマイクロプレート (Corning)
- Microseal B PCRプレートシーリングフィルム (Bio-Rad1)
- SpectraMax i3xマルチモードマイクロプレートリーダー(モレキュラーデバイス)
- SpectraMax M5eマルチモードマイクロプレートリーダー (モレキュラーデバイス)
方法
マイクロプレートリーダー適合性試験
バリデーションキットには、希釈した蛍光色素の個別チューブが含まれていた: FAM、HEX、FAM+HEX。これらはマイクロプレートに直接分注され、熱サイクリングを必要としませんでした。各フルオロフォアのチューブには、HEXとFAMシグナルの標準化を可能にする受動的参照色素であるROXも封入されており、ピペッティングによるばらつきの影響を排除しています。各フルオロフォア(HEX、FAM、HEX+FAM)5 µLを384ウェルプレートのトリプリケートウェルに分注しました。プレートを透明フィルムでシーリングし、560 x gで1分間遠心しました。遠心後、プレートを直ちに M5e および i3x リーダーで表 1 に示すインストゥルメンテーション設定を用いて読み取りました。
KASPジェノタイピング反応の検出
第 2 の検査では、バリデーションキットに既知サンプル 36 検体(DNA サンプル 33 検体およびノー テンプレートコントロール(NTC)3 検体)を封じ込めました。各サンプル5 µLを96ウェルプレートにピペッティングし、PCRを行いました。同量のジェノタイピング混合液(2x KASP Master mix + KASP Assay mix)を各ウェルに添加しました。受動的参照色素ROXはKASP Master mixに含まれていました。プレートを透明フィルムでシーリングし、560 x gで1分間遠心しました。熱サイクリング反応が直ちに開始されました。詳細なサーマルサイクリング条件を表2に示します。反応終了後、各サンプル5μLを384ウェルプレートのウェルに移し、表1に示したインストゥルメンテーションを用いてマイクロプレートリーダーで検出しました。
結果
最初のテストでは、FAM、HEX、両者の混合物を含む各蛍光体の相対蛍光単位をROXに正規化し、正規化値をSoftMax® Proソフトウェアにプロットしました。図1に示すように、3つの異なるクラスタが生成されました。
2番目のテストでは、増幅DNAサンプルのROX規格化蛍光をSoftMax® Proソフトウェアにプロットしました(図2)。3つの明確なクラスタが検出されました。FAMホモ接合体はX軸の近くに、HEXホモ接合体はY軸の近くに位置し、FAMとHEXの両方を封じ込めたヘテロ接合体サンプルは2つのホモ接合体クラスターの間にクラスタリングを形成しました。ノーテンプレート対照は予想通り原点付近にクラスタを形成しました。このデータは、検証キットのベンダーから提供されたジェノタイピング結果とほぼ一致しました。
| インストゥルメンテーション | M5e | i3x |
|---|---|---|
| 読み取りモード | 蛍光 | |
| 読み取りタイプ | エンドポイント測定 | |
| 波長 Lm1(FAM) Lm2 (HEX) Lm3 (ROX) |
485 ex/515 cutoff/520 em 535 ex/550 cutoff/556 em 575 ex/610 cutoff/610 em |
485 ex/520 em 535 ex/556 em 575 ex/610 em |
| PMTおよび光学系 | オート、6閃光/読み取り | 6閃光/読み取り |
表1. データ取得に使用したプレートリーダーの設定。
| ステップ | 説明 | 温度 | 時間 | サイクル数/ステップ |
|---|---|---|---|---|
| 1 | 活性化 | 94°C | 15分 | 1サイクル |
| 2 | 変性 | 94°C | 20秒 | 10サイクル |
| アニーリング/延長 | 61~55°C | 60秒(1サイクルごとに0.6°C降下) | ||
| 3 | 変性 | 94°C | 20 秒 | 30 サイクル |
| アニーリング/延長 | 55°C | 60秒 | ||
表2. KASPジェノタイピング反応の熱サイクル設定
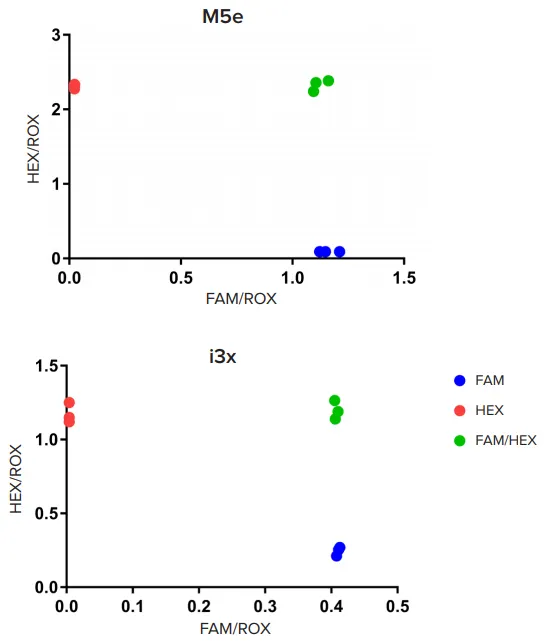
図1. 3種類の蛍光色素を用いたリーダーの検証。M5eおよびi3xの両方から得られたHEX/ROXおよびFAM/ROX値は、3つの特徴的なクラスタを形成しました。
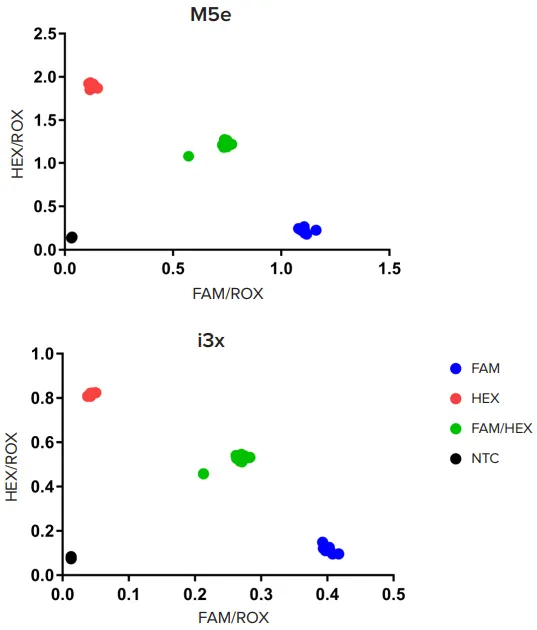
図2. KASPジェノタイピング結果。プレートリーダーで読み取られたPCR産物は、FAMホモ接合体(青)、HEXホモ接合体(赤)、FAM/HEXヘテロ接合体(緑)を含む3つの異なるグループにクラスタリングされました。ノーテンプレートコントロール(NTC)は原点付近にクラスタリングされました。
結論
SpectraMax M5eおよびi3xプレートリーダーは、KASPジェノタイピングアッセイでの使用が完全に検証されています。モノクロメーターベースの光学系は、反応中に存在する3つのジェノタイプすべてを表す蛍光PCR産物を検出するために必要な3つの励起/エミッション波長ペアのセットアップを容易に可能にします。どちらのインストゥルメンテーションでも一貫した結果が得られます。SoftMax® Proソフトウェアによるデータ解析とジェノタイピングにより、正規化された結果を迅速に可視化することができます。
参考文献
1. Chen, X., and Sullivan, P.F. “Single nucleotide polymorphism genotyping: biochemistry, protocol, cost and throughput.” The Pharmacogenomics Journal 3, (2003): 77-96.
2. Patterson, E.L., Fleming, M.B., Kessler, K.C., Nissen, S.J., and Gaines, T.A. "A KASP genotyping method to identify nothern watermilfoil, eurasian watermilfoil and their interspecific hybrid.". Frontiers in Plant Science 8, (2017): 752.
3. Smith, S.M., Maughan, P.J. “SNP genotyping using KASPar assays.” Plant Genotyping 1245, (2015): 243-256.
PDF版(英語)