Application Note SpectraMaxマイクロプレートリーダーを
用いたDNAおよびRNAの吸光度測定
- 標準曲線なしで核酸を直接定量可能
- DNAを250 ng/mLまで定量可能
- SoftMax Proソフトウェアにあらかじめ設定されたプロトコルを搭載
PDF版(英語)
はじめに
モレキュラーデバイスが初めてUV対応のマイクロプレートリーダーを発売した時、マイクロプレートでの紫外線(UV)測定が可能になりました。それ以来、DNA、RNA、タンパク質のマイクロプレート測定は非常にポピュラーになりました。しかしながら、マイクロプレートアッセイでは、特にUV領域で正確な吸光度結果を得るために、マイクロプレート材料の光学特性を認識し、従来のキュベットよりも技術に注意を払う必要があります。このような細部への注意が欠けていることが、アッセイをマイクロプレートに適合させることを困難にする最も頻繁な原因です。
マイクロプレートによる吸光度測定は光路長のばらつきがあり、気液界面での表面効果による干渉を受けやすいです。最新のマイクロプレートリーダーは、旧式のワイドビームプレートリーダーよりも光ビームが小さいため、ホコリによるスプリアスの影響を受けやすいです。読み取り時に光ビームの中に粒子があると、最大0.3 ODのアーティファクト吸光度スパイクを引き起こす可能性があります。したがって、サンプル溶液に粒子がないことが特に重要です。マイクロプレートで正確で再現性のある吸光度結果を得るためには、上記のすべての要因に留意する必要があります。ここでは、SpectraMax®マイクロプレートリーダーでDNA/RNA吸光度測定を最適化するためのガイドラインを示します。
材料と方法
- SpectraMaxマイクロプレートリーダー(UV吸光度検出付き)(モレキュラーデバイス)
- UV透過マイクロプレート
⚪︎使い捨て: コーニングまたはGreiner
⚪︎石英: Hellma
最高品質のDNA吸光度値を得るための推奨事項
- 清潔なマイクロプレートと粒子のない溶液を使用する。バッファーをフィルタリングして(例えばポアサイズ < 5 µm)微粒子を除去し、最良の結果を得るために、使い捨てマイクロプレートを再使用しない。すぐに読み取らない場合は、マイクロプレートに蓋をしてください。
- バッファーブランクの吸光度値を記録します(少なくともトリプリケートでブランクを含める)。ブランク値が期待される範囲に入らない場合は、マイクロプレートの汚れ、ウェル内の微粒子、またはマイクロプレートの欠陥が原因である可能性が最も高いです。使い捨てUVマイクロプレートのプレーティングバックグラウンドOD値は、ロット間でわずかに異なります。新しいロットのきれいな水を満たしたプレートを読み取ることで、予想される平均OD値を決定する必要があります。表 1 に、UV 透明マイクロプレートの OD260 と OD280 の概算値と標準偏差(SD)を示します。コーニング UV プレートとグライナー UV プレートのバックグラウンド OD 値は、石英マイクロプレートとほぼ同等です。
- 低吸収サンプルの場合は、最大光路長(したがって最大吸光度)を得るために最大容量(250~300 µL)を使用します。可能であればデュプリケートまたはトリプリケートも使用します。左右対称のメニスカス形成を促すため、プレートを短時間オートミックスします。
- 少量のサンプル(1~20 µL)と希釈液を使用する場合は、少量のサンプルを最初にウェルにピペッ トし、次に少量のサンプルをウェルにピペッ トします。SpectraMaxインストゥルメンテーションで10~20秒間自動混合し、混合を完了します。少量の試料を最後に添加した場合、混合が不完全なため、結果に大きなばらつきが生じることがあります。
- イオン強度を上げると吸光度が低下することに注意してください *1,*2。DNAを水ではなくTEバッファー生理食塩水に溶解すると、吸光度が約23%低下します(図2)。最高の感度を得るためには、DNAサンプルは脱イオン水に溶かす必要があります。
| OD 260 nm | OD 280 nm | |||
|---|---|---|---|---|
| 平均値 | SD | 平均値 | SD | |
| 石英 | 0.034 | 0.002 | 0.032 | 0.002 |
| コーニングUV* | 0.002 | 0.001 | 0.039 | 0.001 |
| グライナーUV | 0.062 | 0.002 | 0.043 | 0.001 |
表1. 260 nmおよび280 nmにおけるマイクロプレートの光学濃度。値は清浄なプレート5枚の平均値で、各プレートには200 µLの水が満たされています。*コーニングUVプレートでは、波長260 nmの平均値はロット番号により0.046~0.057の範囲でした。
PathCheckを使用して吸光度値を1cmの光路長に正規化する
SpectraMaxマイクロプレートリーダーにはPathCheckテクノロジーが搭載されていて、マイクロプレートの各ウェルの光路長を測定し、吸光度値を1cmの光路長に正規化することで、1cmのキュベットで得られた値と同じにします。
$$ A_{\text{Sample per cm}} = \frac{A_{\text{Sample}}}{\text{pathlength}_{\text{Sample}}\,(\text{cm})} $$
光路長を正規化する前に、マイクロプレート自体の OD を考慮する必要があります。マイクロプレート内のサンプルの生のODは以下の通りです:
$$ OD_{\text{Total}} = A_{\text{Sample}} + A_{\text{Blank}} + OD_{\text{microplate}} $$
ASampleとABlankはどちらも経路長に依存します。ODmicroplateはマイクロプレート材料のODと表面からの反射によるもので、経路長に依存しません。したがって、上記の最初の式で経路長正規化吸光度値を計算する前に、まずこれを差し引く必要があります。SoftMax® Proには、ODmicroplateを減算するための2つのオプションがあります。サンプル量が変動する場合は、オプション1を推奨します。すべてのウェル容量が同じ場合は、ODmicroplateを通常の試薬ブランクに含めることができます(オプション2)。
PathCheckの計算でマイクロプレートのODを除去するオプション
オプション1:プレーティングバックグラウンドODを使用します
(バックグラウンドが低く均一なマイクロプレートに推奨)。
新しいロットの使い捨て UV マイクロプレートごとに、PathCheck で使用する各波長における OD マイクロプレート(「プレートバックグラウンド OD」)を測定します。清浄なプレートにウェルあたり100~200μLの水を入れ(水は190~900nmの間で光をほとんど吸収しないため、量は重要ではありません)、プレートを読み取ります。空気は水と屈折率が異なるため、OD値が異なるので、乾いたプレートは使用しないでください。ある波長における平均ODは、その波長におけるプレートバックグラウンドODです。260 nmと280 nmにおけるODmicroplateの値は、表1に示す値と類似しているはずです。260nmでSDが0.003より大きい場合は、マイクロプレートが汚れているか、欠陥がある可能性があります。
Plate Background ODを決定したら、SoftMax ProのData Reductionダイアログボックスに入力します。通常通り、Template Editorでサンプル、標準品、ブランクの位置を指定し、それに従ってマイクロプレートに注入します。プレートを読み取ると、SoftMax Proは自動的に以下の処理を行います:
- 生のウェル吸光度値からPlate Background ODを差し引きます。
- すべてのサンプル、標準品、ブランクにPathCheckを適用します(Reduction*で選択を解除していない限り)。
- 試薬ブランクを差し引きます。
*PathCheckの選択を解除すると、プレートのバックグラウンド減算も解除されます。
オプション 2: ODマイクロプレートをプレートブランクの一部に含める
すべてのウェルの容量(およびパスの長さ)が同じで、すべてのサンプルが同じ溶液でブランクされている場合に、このオプションを使用します。
設計上、SoftMax Proは経路長の正規化後にプレートブランク(試薬ブランク)を差し引きます。ODmicroplateは、すべてのサンプル量とブランク量が同一である場合(およびその場合のみ)に、プレートブランクの構成要素として差し引くことができます。
マイクロプレート内の3つ以上のウェルを選択し、ブランキング溶液(通常は水またはバッファー)を封入します。SoftMax ProのTemplate Editorで、これらのウェルを "Plate Blank "**として割り当てます。残りのウェルをサンプルおよび標準液として指定し、それに従ってプレートを準備します。プレートを読み取ると、SoftMax Proは自動的に以下の処理を行います:
- すべてのサンプルとブランクにPathCheckを適用します(選択を解除した場合を除く)。
- マイクロプレートの各ウェルからブランクの平均値を差し引きます。すべてのサンプルとブランクのパス長が同一であることを前提に、ODmicroplateにパス長正規化を適用する際の潜在的なエラーはキャンセルされます。
**SoftMax Proは、マイクロプレートの各ウェルからプレー トブランクウェルの平均吸光度値を差し引きます。
SpectraMaxリーダーでのDNA測定例
子牛胸腺 DNA のアリコート(水中 \( \frac{0.1\text{ー}2.0\,\mu\text{g}}{\text{mL}} \))を、PathCheck を適用した Corning UV マイクロプレートで 260 nm で読み取りました(図 1)。計算上の検出限界(ブランク値のゼロ基準に対して、正の標準偏差(SD)3個分を超える吸光度を示す量)は約\( \frac{25\,\text{ng}}{\text{well}} \)でした。非常に保守的な基準(試薬ブランクのゼロ値を基準として、正の標準偏差(SD)10個分を超える吸光度値を示す量)を用いて計算した定量限界は、約\( \frac{75\,\text{ng}}{\text{well}} \)でした。300μLを封じ込めたウェルでは、この限界値は\( \frac{250\,\text{ng}}{\text{mL}} \)となります。より低い限界値は、より控えめな基準で達成できますが、技術に厳密な注意を払う必要があります。上記の結果は、最適条件下であらかじめ希釈した溶液を用いて得られたものです。その代わりに、各ウェルに少量の犠牲DNAサンプルと希釈液を入れるプロトコールでは、定量限界は多少高くなります。
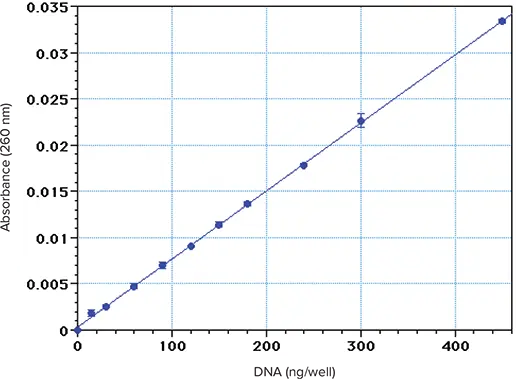
図1. 子牛胸腺DNAを水に溶解して得られたスタンダードカーブ。トリプリケートした 300 µL のアリコートを Costar UV マイクロプレートに入れ、PathCheck を適用して 260 nm で読み取りました。
マイクロプレートの結果は、分光光度計のA260測定によるDNAの検出限界とよく一致しています *3,*4。これは、入手可能なUV透過マイクロプレートが高品質であること、マイクロプレートウェル全体の光路長が約1cmであることを考慮すると、予想外の結果ではないです。DNA濃度が低い試料を封じ込めた場合、UV吸光度では測定できず、蛍光色素との反応など、別の手法でしか測定できないです。
吸光度を用いたDNA/RNAの推定
DNA/RNA濃度は、一般的にA260値を1cm吸光度の値で割る(またはその逆数を掛ける)ことで推定されます。PathCheckを使用すると、SpectraMaxプレートリーダーは自動的にサンプルの吸光度値を1cmの光路長に正規化し、濃度を計算することができます。260nmにおける二本鎖DNAの\( \frac{1}{\text{absorptivity}} \)の値は、一般に1cmの光路長に対して\( \frac{\sim 50\,\mu\text{g}}{\text{mL}} \)と仮定されています *3,*4。
しかし、この値が正しいのは、溶液の塩濃度が比較的高い場合に限られるようです(図2)。DNAはTEバッファー中では水よりも吸光度が15%低く、TE+生理食塩水(TES)中では23%低いです。\( \frac{1}{\text{absorptivity}} \) の計算値は、水、TE、TES中のDNAについて、それぞれ\( \frac{38\,\mu\text{g}}{\text{mL}} \)、\( \frac{45\,\mu\text{g}}{\text{mL}} \)、\( \frac{50\,\mu\text{g}}{\text{mL}} \) でした(図2の挿入図)。
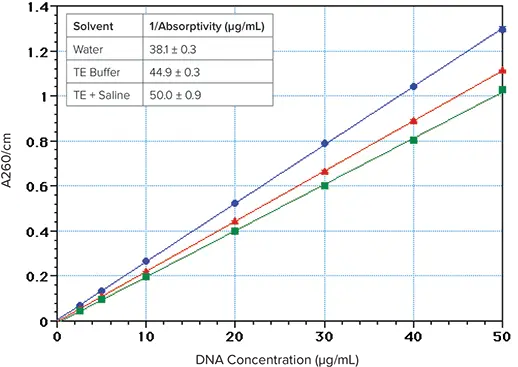
図2. 260nmのDNA吸収に対するイオン強度の影響。DNA(Sigma Type I "Highly polymerized"、Cat.No.1501)を脱イオン水(青丸)またはTEバッファー(10 mM Tris, 1 mM EDTA, pH 7.4)(赤三角)またはTES(TEバッファー\( \scriptstyle{+\,0.9\%\,\text{NaCl}} \))(緑四角)に溶解した。挿入図:\( \frac{1}{\text{absorptivity}} \);7濃度(\( \frac{2.5\,\mu\text{g}}{\text{mL}} \text{~} \frac{50\,\mu\text{g}}{\text{mL}} \))の平均、各4反復。
SoftMax Proを使用したDNA濃度の計算
DNA/RNAアッセイプロトコルでは、作業可能なサンプル量を得るために希釈剤を加えた少量(2~10 µL)サンプルのA260測定を行うことがよくあります。例えば、シークエンシングやその他のアッセイに先立ち、血液や組織サンプルからDNAを抽出する場合です。アッセイに進む前に、抽出液の少量の犠牲アリコートのDNA濃度を測定し、その結果を使用して、所望のDNA量をアッセイに移すために必要なアリコートのサイズを計算します。SoftMax Proは、SpectraMaxリーダーからのA260値を自動的に正規化し、濃度を決定し、目的のアリコートサイズを計算することができます。SoftMax Proは、0.5 µL単位で設定可能なリキッドハンドリングシステムに対応するため、0.5 µL単位で四捨五入することもできます。図3は、犠牲となる分注量が5 µLで、アッセイの目標量が0.4 µgの場合の例です。各サンプルについて公称分注量が計算され、その量も0.5 µL単位で四捨五入されました。これらの計算結果は、外部のデータ処理システムで使用するために簡単にエクスポートできます。
| DNA抽出物 | |||||
|---|---|---|---|---|---|
| サンプル | ウェル | OD 260 nm | ng/μL | ターゲット | アリコート |
| 1 |
D1 D2 |
0.125 0.126 |
250.9 | 1.59 | 1.5 |
| 2 |
E1 E2 |
0.074 0.071 |
144.4 | 2.77 | 3.0 |
| 3 |
F1 F2 |
0.120 0.140 |
259.5 | 1.54 | 1.5 |
| 4 |
G1 G2 |
0.048 0.050 |
98.2 | 4.07 | 4.0 |
| 5 |
H1 H2 |
0.142 0.149 |
290.9 | 1.37 | 1.5 |
*400ngを移動するのに必要なアリクオットサイズ
**アリコート量は0.5 µL未満を四捨五入。
図3. DNA抽出物。SoftMax Proで作成されたレポートの例。DNA抽出物の結果と、アッセイへの移し替えなど、指定した量のDNAを封じ込めるために必要な分注量の計算結果が記載されています。
結論
技術に注意すれば、正確で再現性のあるDNA/RNA測定をマイクロプレートで簡単に行うことができます。定量下限値は、従来のUV-VIS分光光度計で得られた値に匹敵します。測定結果は、PathCheckを使用して1cmの光路長に自動的に正規化することができ、標準的な1cmの光路長のキュベットで得られた値と同等の値が得られます。SoftMax Proは、特異性のある量のDNA/RNAを封じ込めるのに必要な分注量を計算し、結果をカスタマイズしたウェルフォーマットで報告することができます。SoftMax Proには、吸光度ベースの核酸定量法用にあらかじめ設定されたプロトコルがあり、データの作成と解析が容易です。
参考文献
- Beaven, G.H., E.R. Holiday and E.A. Johnson. 1955. Optical properties of nucleic acids and their components, p. 493–553. The Nucleic Acids, Vol. 1. Academic Press, New York. (E. Chargaff and J.N. Davidson, Eds.)
- Wilfinger, W.W., K. Mackey and P. Chomczynski. 1997. Effect of pH and Ionic strength on the spectrophotometric assessment of nucleic acid purity. BioTechniques 22: 474–479.
- Sambrook, J., E. Fritsch and T. Maniatis. 1989. Molecular Cloning: A Laboratory Manual, 2nd ed. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York. p. E.5.
- Gallagher, S.R. 1994. Quantitation of DNA and RNA with Absorption and Fluorescence Spectroscopy. Current Protocols In Molecular Biolgy, Vol. 3. John Wiley & Sons, pp. A3.D.1–A3.D3.
SpectraMaxマイクロプレートリーダーについて詳しくはこちら >>
PDF版(英語)