Application Note GeneBLAzer 細胞ベース FRET アッセイで
GPCR 活性を評価する方法
- デュアルPMTによる高速リードタイムとFRETシグナル精度の向上
- 高感度を実現する最適化されたGeneBLAzer検出カートリッジ
- 結果を損なわない迅速なリードを可能にするオンザフライスピード
PDF版(英語)
Cathleen Salomo|アプリケーションサイエンティスト|モレキュラーデバイス
はじめに
このアプリケーションノートでは、SpectraMax® Paradigm® マルチモードマイクロプレートリーダー上でのLife Technologies GeneBLAzerアッセイの使用について説明します。最適な解析条件は、ブラックウォール・クリアボトムプレートを用いたさまざまなプレートリーダー設定で評価しました。
GeneBLAzerアッセイは、蛍光共鳴エネルギー移動(FRET)に基づく手法であり、Gタンパク質共役受容体(GPCR)やその他の創薬ターゲットをin vivo(生細胞)またはin vitro(細胞溶解液)で研究するために使用されます。GeneBLAzerテクノロジーは、哺乳類細胞内でのβ-ラクタマーゼレポーター活性に基づいており、FRETベースの基質(CCF2またはCCF4)を介して検出されます。この基質には、クマリンとフルオレセインという2種類の蛍光色素が含まれています(図1)。β-ラクタマーゼが発現していない場合、基質はそのまま保持されます。この状態では、クマリンが励起され、FRETが起こり、エネルギーがフルオレセインに移動し、フルオレセインが励起されて緑色の光を発します。しかし、β-ラクタマーゼが発現すると、基質が切断され、2つの蛍光色素が分離し、エネルギー移動が阻害されます。この場合、クマリンのみが励起され、青色の光を発します。レポーター応答は青:緑比として表されます。この正規化された比率測定は、実験ノイズを最小限に抑え、細胞数やトランスフェクション効率の変動などの誤差に対して頑健です。

図1. GeneBLAzerテクノロジーの仕組み
CCF-2基質にはクマリンとフルオレセインが含まれています。基質が intact の場合、クマリンの励起によりFRETがフルオレセインに起こり、緑色の光を発します。β-ラクタマーゼ(BLA)が発現すると、基質が切断され、エネルギー移動が阻害され、クマリンの励起によって青色蛍光シグナルが生じます。結果として得られる青:緑比は、正規化されたレポーター応答を提供します。
SpectraMax Paradigm プレートリーダーは、FRETシグナルの高速読み取りと精度向上のためにデュアル光電子増倍管(PMT)を搭載しています。ユーザーアップグレード可能な設計により、光学検出カートリッジをニーズに応じて構成できます。これには、アッセイに柔軟なマルチモードカートリッジやアッセイ専用カートリッジが含まれます。蛍光強度(FI)GeneBLAzer検出カートリッジは、InvitrogenのGeneBLAzerアッセイ用に開発され、FRETドナー部分の励起に超高出力LEDと最適化フィルターセットを組み合わせ、優れた感度を提供します。さらに、HTS(ハイスループットスクリーニング)用途に対応するため、リーダーは最大1536ウェルマイクロプレートフォーマットをサポートし、オプションのStakMax® マイクロプレートスタッカーや他のロボットプラットフォームと統合可能です。オンザフライ読み取りにより、データ品質を維持しながらプレート読み取り時間を短縮できます。
材料と方法
アッセイ準備と機器設定
**GeneBLAzer M1 CHO-K1 DA細胞(ThermoFisher, K1365)**は、Gタンパク質活性化を刺激するためにカルバコール(カルバモイルコリン塩化物、Sigma C4382-10G)で処理しました(図2)。活性化ステージは、LiveBLAzer-FRET B/G Loading Kit(ThermoFisher, K1095; CCF4-AM基質を使用)を用いてSpectraMax Paradigmリーダーで測定しました。最適化されたプレートリーダー設定は表1に示します。

図2. GeneBLAzer CHO-K1 DA細胞におけるGタンパク質活性化の刺激 細胞は右から左へ、カルバコールの濃度を増加させて刺激しました。
| パラメーター | 設定 |
|---|---|
| 検出カートリッジ | 蛍光強度(FI) GeneBLAzer検出カートリッジ(品番:0200-7006) |
| フィルター:波長幅 |
EX:406-15 EM1: 465-35 EM2: 535-25 |
| 光源 | LED、超高出力 |
| リードモード | FRET |
| リードタイプ | エンドポイント、ボトムリード |
| PMTおよび光学系 |
オプション1:ウェルあたりの積分時間140msでストップ&ゴー オプション2:オンザフライ性能 オプション3:オンザフライスピード |
| リード高さ | Z-height最適化ツールを使用して、Plateタイプごとに最適化。 |
表1. GeneBLAzer検出カートリッジを搭載したSpectraMax Paradigmプレートリーダーの機器設定
データ解析
データおよびカーブフィット解析はSoftMax® Proソフトウェアを使用して実施しました。青:緑比およびZ´因子の計算は以下の式で求めました:
\[ \text{Ratio} = \frac{\text{(signal at }465\,\text{nm} - \text{background at }465\,\text{nm)}} {\text{(signal at }535\,\text{nm} - \text{background at }535\,\text{nm)}} \]
\[ Z^{\prime}\ \text{factor} = 1 - \frac{3\bigl(\hat{\sigma}_{p} + \hat{\sigma}_{n}\bigr)} {\left|\hat{\mu}_{p} - \hat{\mu}_{n}\right|} \]
pはポジティブコントロール、nはネガティブコントロールを示します。
結果
プレートタイプの比較
アッセイ性能を比較するため、細胞をさまざまなプレート素材およびウェル密度で培養しました。プレートの特性と詳細は表2にまとめています。すべてのマイクロプレートでEC₅₀値は同程度であり、Z´因子は0.8以上を達成しました(図3)。最も広いアッセイウィンドウを示した用量反応曲線は、1536ウェルSCREENSTARマイクロプレートで観察されました。標準的な組織培養処理(CELLSTAR)は、使用したトランスフェクトCHO細胞の接着と増殖に十分でした。SpectraMax® ParadigmリーダーとGeneBLAzer検出カートリッジは、弱いFRETシグナルでさえ検出できるほど高感度であり、GeneBLAzerアッセイを1536ウェルフォーマットに小型化しても性能を損なうことはありませんでした。384ウェルおよび1536ウェルプレートの両方で同等の結果が得られ、本研究で使用した細胞株およびGeneBLAzer試薬に適用可能です。
| プレート名 | ソース | プレート |
|---|---|---|
|
μクリア 384 ウェル # 781 091 1536-well # 783 092 |
Greiner Bio-One GmbH | ポリスチレン(PS)マイクロプレート(薄いPSフィルム底付き |
|
スクリーンスター 384ウェル # 789836 1536ウェル # 789866 |
Greiner Bio-One GmbH | シクロオレフィンマイクロプレート、190μmの薄いシクロオレフィンフィルム底付き。 |
表2. 使用したプレートタイプ

図3. 異なるプレートタイプを用いた結果比較 データは4パラメータ(対数)曲線でフィッティングしました。
リード速度の比較
SpectraMax Paradigmプレートリーダーは、3種類のリード速度オプションを提供します。1536ウェルμClearプレートを用いて、EC₅₀およびZ´因子を維持しながら最速のリード時間を決定しました。
プレートリーダーで利用可能なリード速度は以下の通りです:
- Stop and go:プレートが停止し、ウェル中央がリードヘッド下に位置する間にシグナルを検出。シグナル積分時間はユーザー設定可能。本実験ではウェルあたり140 msに設定。
- On-the-fly:プレート移動中にウェル中央領域のシグナルを検出。積分時間は固定。
1. Optimized for speed:最速のリード時間を得るために設計。
2. Optimized for performance:品質向上のためリード速度を低下。
結果は図4にまとめています。異なるリードモードでのEC₅₀値およびZ´因子は同等でした。したがって、本アプリケーションでは最速のリード速度であるOn-the-fly optimized for speedを使用し、プレートリード時間を最小化して迅速に結果を得ることが可能です。
| Stop and go | On-the-flyパフォーマンス | On-the-flyスピード | |
|---|---|---|---|
|
時間 プレート合計 |
6分 | 1.5分 | 1分 |
|
リードモード パターン 積算時間/ウェル |
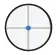 140ms |
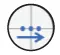 |
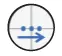 |
|
EC50 |
0.164 | 0.161 | 0.161 |
|
Z' prime |
0.84 | 0.79 | 0.85 |
図4. SpectraMax Paradigmプレートリーダーによる1536ウェルプレートのリード速度比較 データは4パラメータ(対数)曲線でフィッティングしました。
結論
このアプリケーションノートは、SpectraMax ParadigmプレートリーダーでのGeneBLAzerアッセイの優れた性能を示しています。アッセイは1536ウェルフォーマットに小型化でき、最速のリード速度を使用することで、1536ウェルプレートの総リード時間をわずか1分に短縮しながら結果の精度を損なうことはありません。
参考文献
Zhang, et al., J Biomol Screen, April 1999 Volume 4 No. 2, pp. 67-73.
PDF版(英語)
