Application Note β-ラクタマーゼ阻害剤とタンパク質の結合親和性: FlexStation 3 マイクロプレートリーダーを用いた
TEM-1酵素によるβ-ラクタム基質の加水分解および阻害剤結合の検出
PDF版(英語)
Melinda Hanes, Tracy Handel, UCSD School of Pharmacy and Pharmaceutical Sciences, La Jolla, CA,
and Anita Kant, モレキュラーデバイス, 1311 Orleans Drive, Sunnyvale, CA 94089.

イントロダクション
β-ラクタム系抗生物質に対する細菌の耐性は、クラスA β-ラクタム分解酵素の産生を通じて増加しており、医療界で深刻な問題となっています *1。土壌細菌 Streptomyces clavuligeris が産生する β-ラクタマーゼ阻害タンパク質(BLIP)は、幅広い親和性で複数のクラス A β-ラクタマーゼを阻害し、これにより命を救う抗生物質の有効性をリストアさせます *2。BLIPの結合パートナーとして知られているのは、大腸菌TEM-1、セラチア菌SME-1、炭疽菌BlaI(これら3つすべてに対してナノモル親和性)、肺炎桿菌SHV-1(マイクロモル親和性)、プロテウス菌K1(ピコモル親和性)などです(2-4)。生物製剤の相互作用は生体系に普遍的に存在するにもかかわらず、タンパク質相互作用の親和性と特異性の生化学的基盤を支配する法則はほとんど存在しません。阻害を成功させるには、どのような相互作用が十分で必要なのでしょうか? 様々な酵素を認識し阻害するBLIPの能力に関する洞察は、新治療薬の開発に直ちに役立ちます。変異誘発データはこの洞察を得るために非常に貴重です。
標的変異誘発実験のための結合親和性を決定するハイスループットな方法は、私たちの研究を大いに助けてくれます。また、タンパク質や基質などの試薬の使用量を減らすことができる、少量のサンプルを使用できる能力も重要です。これは、タンパク質が貴重な場合に特に重要です。例えば、親和性の低い変異体では、必要な阻害剤の量は、タイトバインディング阻害剤で同じ反応を得るのに必要な量よりはるかに多いです。
モレキュラーデバイスの FlexStation® 3 システムは、分注モジュール付 きマルチモードマイクロプレートリーダーです。分注モジュールは、ソースプレートからアッセイプレートへ試薬を伝達するマルチチャンネルピペッティングを備えています(図1参照)。(図1参照)マイクロプレートは下方から測定を行うため、試薬がアッセイプレートに添加されると同時にカイネティック測定データを高速で収集できる「添加と測定」の同時実行が可能です。このピペッターには、96ウェルプレートまたは384ウェルプレートにそれぞれ適合する8チャンネルまたは16チャンネルフォーマットがあります。この "Flex "モードでは、一度に一つのカラムをアッセイします。このフォーマットは、一度に1ウェルしか評価できないインジェクターベースのシステムと比較して、スループットを最大8倍または16倍に増加させることができます。ピペッターが試薬を化合物プレートのウェルからアッセイプレートのウェルに移送するため、試薬のクロスコンタミネーションを防ぐことができます。システムは前処理を必要としないため、貴重な化合物のロスを最小限に抑えることができます。
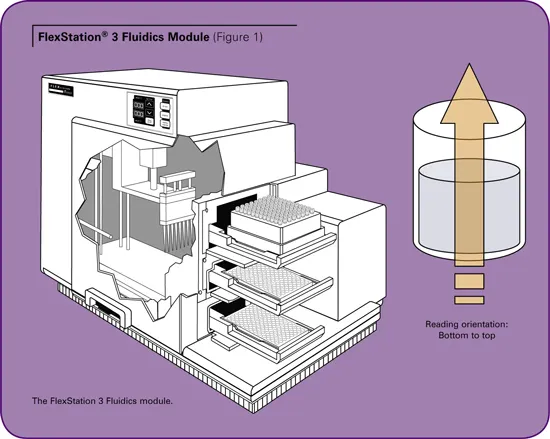
このアッセイでは、TEM-1をアッセイプレート中でBLIPと混合し、比色分析基質であるニトロセフィンをピペッティングで添加することにより反応を開始させます。反応は直ちに進行します。反応速度は、486nmの吸光度を3分間測定することによりモニターされます。反応の初期速度は結合親和性の算出に使用されます。FlexStation® 3システムは、この測定をリアルタイムで容易にします。吸光度は透過現象であり、光源と読み取りヘッド がサンプルの反対側にある必要があります(図1参照)。リードヘッドとピペッターの衝突を避けるため、 リードヘッドが最初のベースライン読み取りを行い、 脇に移動します。その後、ピペッターが移動して分注を行い、分注が完了したら離れます。その後、リードヘッドが読み取りを再開します。
ここでは、FlexStation 3システムを用いてBLIPのTEM-1に対する結合親和性(0.96 ± 0.08 nM)を測定し、文献に報告されている他の方法(0.2~2 nM)と同様の結果が得られたことを報告します(*2, *5-*7).
材料
- FlexStation® 3 マルチモードマイクロプレートリーダー (モレキュラーデバイス)
- β-ラクタマーゼ阻害タンパク質(BLIP)
- TEM-1 ß-ラクタマーゼ、PBS、BSA (NEB)
- ニトロセフィン (Calbiochem)
- 96ウェルアッセイプレート(Costar)
方法
タンパク質の調製
TEM-1をOmpA分泌シグナルの後ろのpET-24a(+) (Novagen)にクローニングしました;ネイティブ分泌シグナルを含むBLIPをpET-26b(+) (Novagen)にクローニングしました。両タンパク質は大腸菌BL21(DE3)(Stratagene)でIPTG誘導により3時間発現させました。浸透圧ショックで採取したペリプラズム画分を、陰イオン交換クロマトグラフィー、TEM-1ではDEAE-セファロース、BLIPではQ-セファロース(GEヘルスケア)、続いてSuperdex75カラム(GEヘルスケア)を用いたサイズ排除クロマトグラフィーでさらに精製しました。
アッセイセットアップ
適量のTEM-1、BLIP、およびBSAを、96ウェルプレートに三重測定した総容量90μLのPBS中で混合し、蓋をして2時間インキュベートしました。最終基質濃度121 µMの10Xニトロセフィンを10 µL添加した時点で、Flex read typeを用いて反応が開始しました。
最終反応条件 2 nM TEM-1、100 µg/ mL BSA、121 µM ニトロセフィン、全量100 µLのPBS中、BLIP濃度を変化させました(0、1、2、4、8、16、32、64 nM)。
機器設定:
反応混合物の吸光度を測定するため、機器はFlex readタイプに設定しました。
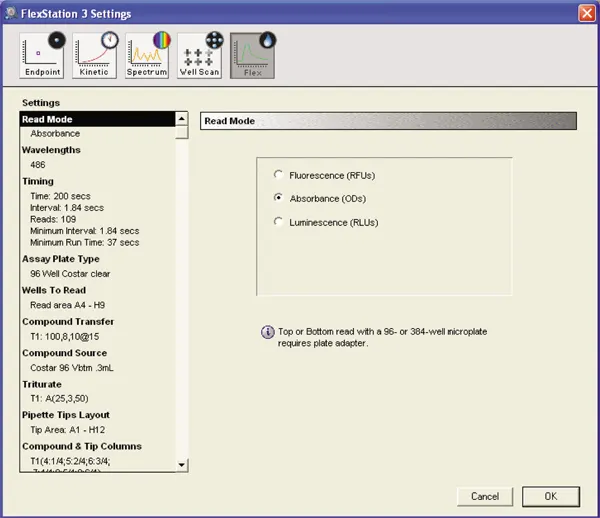
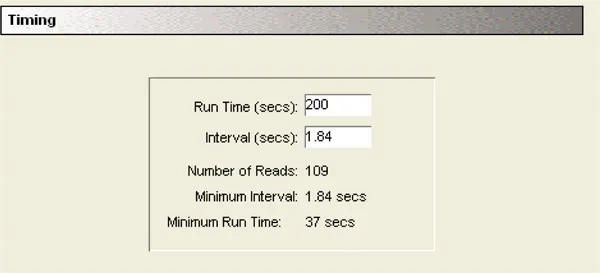
カイネティックの設定:
各カラムは、読み取りヘッドがウェルAからHに移動し、次の読み取りのためにAに戻る間、200秒間読み取られた。ウェル内の2つの読み取り間の最小時間間隔は1.84秒であった。
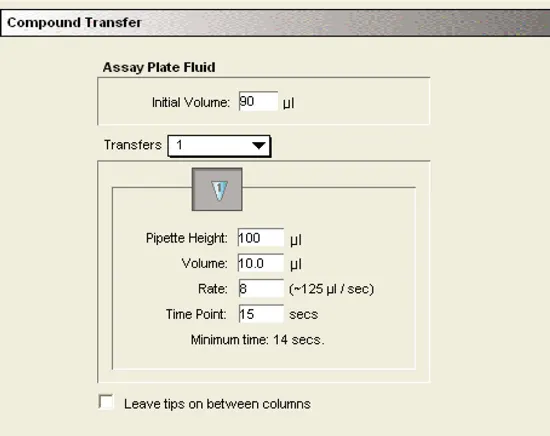
液体移動の設定:
TEM-1とBLIPを封じ込めた初期容量は90μLでした。10µLのニトロセフィン基質を、チップとウェル内の液体が接触しないように、100µLの高さでピペッターで加えました。
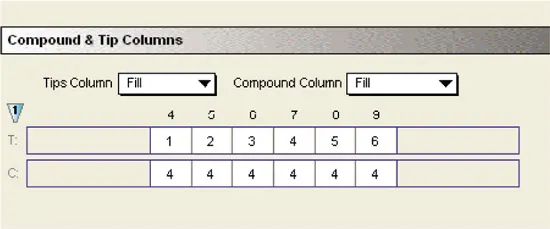
化合物とチップ
アッセイプレートの異なるカラムに基質を分注するため、別々のチップを使用しました。基質は化合物プレートの単一カラムからピッキングしました。チップと化合物のカラムを柔軟に選択できるため、チップと化合物を効率的かつ経済的に使用できました。
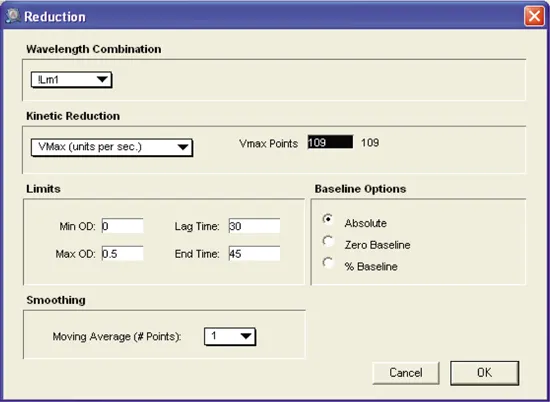
Reductionの設定:
反応の初期速度は、カイネティック反応オプションとして「VMax」を選択して計算しました。さらに、30秒から45秒の間の適切な時間間隔を選択することで、加水分解反応の線形位相に制限しました。
結果
2 nMのTEM-1、BLIP(0-64 nM)および100 µg/mL BSAを、96ウェルプレートに三重測定で全量90 µLのPBS中で混合し、蓋をして2時間インキュベートしました。最終基質濃度が121 µMになるように10 µLの10Xニトロセフィンを添加すると、Flex read typeで反応が開始しました。一度に一つのカラムを評価しました。図3はカラム6の結果を示しています。
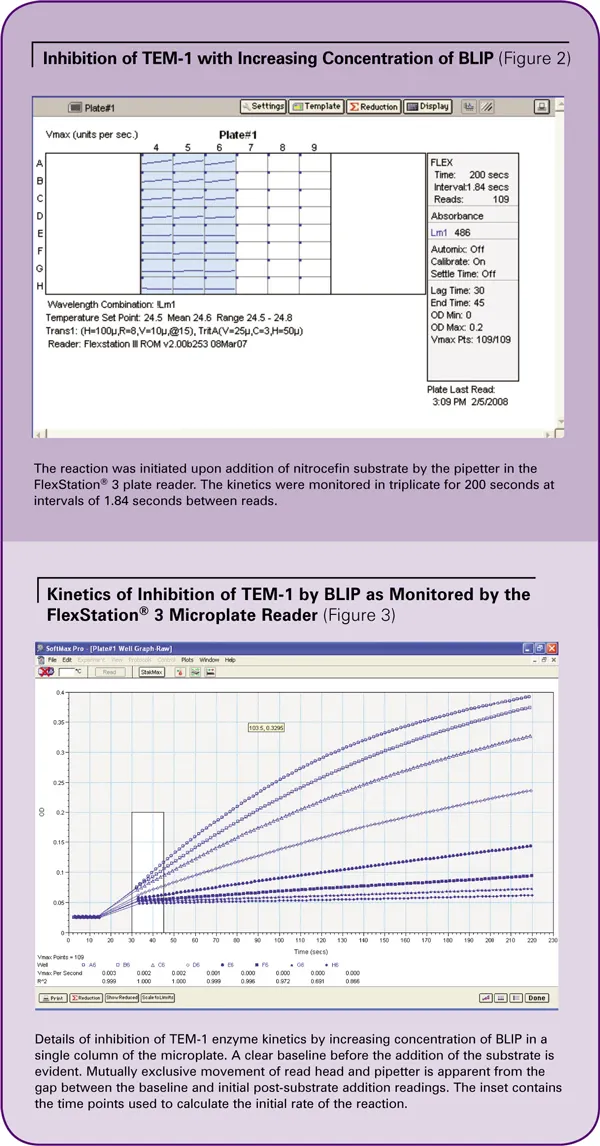
ウェルA6からH6には0、1、2、4、8、16、32、64nMのBLIPを封じ込めた。反応はリアルタイムで観察されました。ベースラインは基質添加前に記録しました。ギャップは、読み取りヘッドがプレートから離れる動きと、ピペッターによる基質の分注を反映しています。基質が分注された後、リードヘッドは元の位置に戻り、反応を検出し続けました。
BLIPの濃度を上げると、TEM-1の阻害が明らかになりました。反応の初期速度は、図3のカイネティックトレースのハイライト部分で示されるように、30秒から45秒の間で計算されました。
データ解析
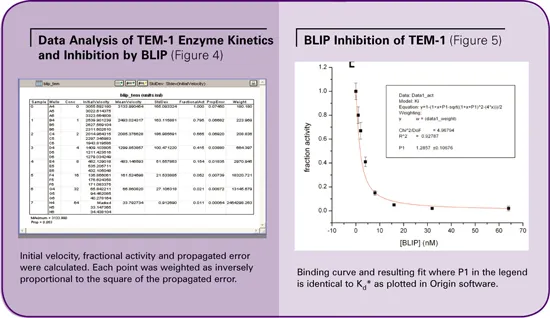
反応の初期速度は、SoftMax® Proソフトウェアによって、加水分解の線形位相における曲線の傾きに基づいて計算されました(図3のVMax単位/秒)。平均速度と標準偏差を算出しました。部分活性は、封じ込め剤を含まないサンプルに対して正規化することで算出しました。(図4参照)。
部分活性と伝播誤差に対応するデータ列は、さらなる分析のためにOriginソフトウェアに手動で転送されました。分画活性は阻害剤の濃度に対してプロットされ、Originを使って以下の式に当てはめました。
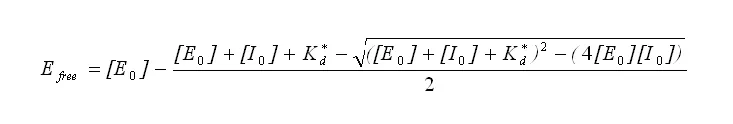
ここで、Efreeは活性から算出した残存遊離酵素濃度、[E0]は初期酵素濃度、[I0]はBLIP濃度、Kd*は見かけの平衡定数です。Kd*は基質の存在により次式で補正しました。[S]は基質濃度、Kmは基質に対する酵素の親和性です。
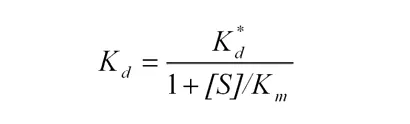
TEM-1に対するBLIPの結合親和性は0.96±0.08 nMであり、他の様々な方法で測定した0.2~2 nMの文献値と一致した(図5参照)。(図5参照)。
結論
FlexStation® 3 マルチモードマイクロプレートリー ダーを用いると、β-ラクタマーゼと阻害タンパク質、 TEM-1 および BLIP の結合親和性を定量的に測定でき ることが、他の方法と比較して明らかになりました。FlexStation 3システムは、貴重な試薬と生物学的材料を経済的に使用し、複数の変異体と阻害剤をリアルタイムで 研究するハイスループット・プラットフォームをご提供します。
参考文献
- .Perez, F., et al., The continuing challenge of ESBLs. Current Opinion in Pharmacology, 2007. 7(5): p. 459-469.
- Strynadka, N.C.J., et al., Structural and kinetic characterization of a [ß]-lactamaseinhibitor protein. Nature, 1994. 368(6472): p. 657-660.
- Zhang, Z. and T. Palzkill, Determinants of Binding Affinity and Specificity for the Interaction of TEM-1 and SME-1 {ß}-Lactamase with {ß}-Lactamase Inhibitory Protein. J Biol Chem, 2003. 278(46): p. 45706-45712.
- Zhang, Z. and T. Palzkill, Dissecting the Protein-Protein Interface between {ß}-Lactamase Inhibitory Protein and Class A {ß}-Lactamases. J Biol Chem, 2004. 279(41): p. 42860-42866.
- Rudgers, G.W. and T. Palzkill, Identification of Residues in ß -Lactamase Critical for Binding ß-Lactamase Inhibitory Protein. J Biol Chem, 1999. 274(11): p. 6963-6971.
- Albeck, S. and G. Schreiber, Biophysical Characterization of the Interaction of the B2;- Lactamase TEM-1 with Its Protein Inhibitor BLIP. Biochemistry, 1999. 38(1): p. 11-21.
- Petrosino, J., et al., Contributions of Aspartate 49 and Phenylalanine 142 Residues of a Tight Binding Inhibitory Protein of ß-Lactamases. J Biol Chem, 1999. 274(4): p. 2394-2400.
FlexStation 3 マルチモードマイクロプレートリーダーの詳細はこちら >>
PDF版(英語)