Application Note IPTGによるタンパク質発現と
細胞増殖のマルチタスク動態測定
- 複数の異なるシグナルを時間経過とともに同時に測定します
- 包括的なデータ解析のために、多様なアルゴリズムとカーブフィットを活用します
- Workflow Editorを使用して、シンプルなプロトコールを設定します
PDF版(英語)
はじめに
複数の異なるシグナル出力を時間経過とともに同時に測定することは、タンパク質や化合物が細胞増殖や遺伝子発現に与える影響を研究するうえで特に有用です。本テクニカルノートでは、SoftMax® Pro 7ソフトウェアを使用して、細胞増殖(吸光度)とタンパク質発現(蛍光)を測定します。
細菌において、lacオペロンは1つのプロモーターにより制御される遺伝子群であり、ß-ガラクトシダーゼをコードします。タンパク質の配列をlacオペロンを含むプラスミドに連結し、そのプラスミドで細菌をトランスフェクトすると、目的のタンパク質を発現させることができます。通常、lacプロモーターはアロステリックに抑制されていますが、イソプロピル ß-D-1-チオガラクトピラノシド(IPTG)が存在すると、リプレッサーがプロモーター配列から解離し、目的のタンパク質が発現します。逆に、IPTGが過剰に存在すると、細胞はタンパク質発現に過剰な細胞資源を割き、その結果、増殖速度が低下します *1。
本研究では、IPTGの希釈系列に対するE. coliの応答を、細胞密度とタンパク質発現を同時に測定することで示します。
材料
- SpectraMax® M2マルチモードマイクロプレートリーダー(モレキュラーデバイス)
- ルリアブロス(LB)培地(Teknova)
- アンピシリンナトリウム塩(Teknova)
- 1 M IPTG(Teknova)
- 96ウェル透明平底ポリスチレンマイクロプレート (Greiner)
- pBbE5-RFPプラスミドを封じ込めた大腸菌 (Keasling Lab)
方法
pBbE5-RFPプラスミド(図1)を含むE. coliは、Jay D. Keasling教授より提供されました *2。E. coli株は、100 µMアンピシリンを含むLB培地でOD600が0.3になるまで培養しました。その後、100 µLのE. coliをクリアな96ウェルマイクロプレートに移しました。IPTGは500 µMから開始する2倍段階希釈系列でE. coliに処理しました。0 µM IPTGおよび培地のみのコントロールもアッセイしました。
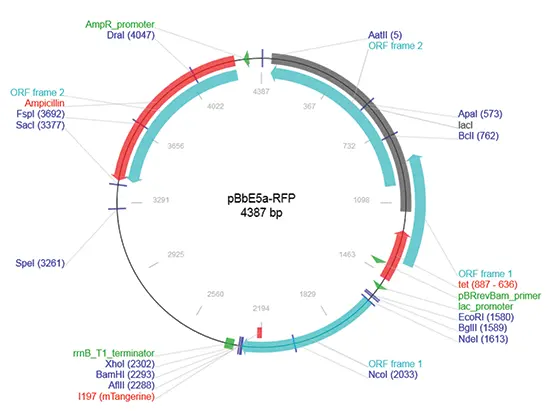
図1. pBbE5a-RFPプラスミドマップ。
マイクロプレートはSpectraMax M2マルチモードマイクロプレートリーダーにセットし、動態アッセイの間32°Cでインキュベートしました。SoftMax Pro 7 Workflow Editorを使用して、吸光度測定と蛍光測定を含む動態サイクルを作成し、測定間に10秒間のプレートシェイクを設定しました。設定は表1に示します。動態サイクルは10分ごとに繰り返し、合計24時間測定しました。得られたデュアルリードモードの動態データはSoftMax Pro 7ソフトウェアで解析しました。
| Read mode | ||
|---|---|---|
| Absorbance | Fluorescence | |
| Wavelength (nm) | 600 | Ex/Em = 530/625 |
| Read type | Bottom read | Bottom read |
| Interval | 1 read/10 minutes | 1 read/10 minutes |
| Optical settings | - | 10 flashes/read |
| PMT gain | - | High |
表1. 細胞密度とタンパク質発現を測定するための装置設定。
測定結果
吸光度と蛍光測定の動態トレースは、それぞれ図2と図3に示します。図2では、IPTG濃度の増加に伴いOD600値が低下し、細菌増殖に悪影響を与えることが示されました。図3では、IPTG濃度の増加によって赤色蛍光タンパク質(RFP)の発現が増加することはなく、逆に全体的なRFP発現が減少しました。この減少は、細菌増殖のプラトー化によるものと考えられます。
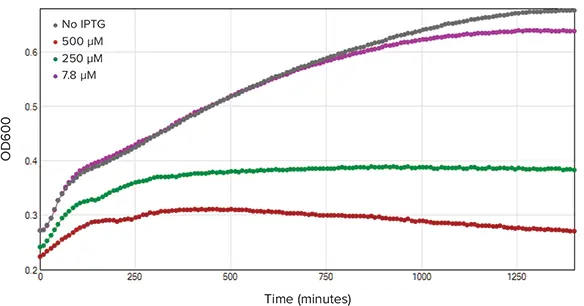
図2. OD600動態トレース。OD600測定は24時間にわたり10分ごとに記録しました(n=3)。IPTG濃度が増加すると、細菌の増殖速度は低下しました。
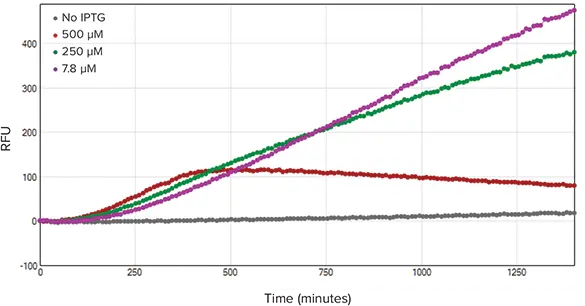
図3. 蛍光動態トレース。蛍光はOD600測定と同時に記録しました(n=3)。500 µM IPTG処理ウェルを除き、蛍光出力はすべての濃度範囲でほぼ同様でした。
図4では、吸光度と蛍光の両方のデータセットに対して積分を適用し、曲線下面積(AUC)を算出しました。この解析はSoftMax Pro 7ソフトウェアで実施し、IPTGが細胞増殖とタンパク質発現に与える影響を要約しています。蛍光に対するIPTGの影響は、500 µMを除き、すべての濃度でほぼ同様でしたが、IPTG濃度の増加により細菌増殖(吸光度)は低下しました。
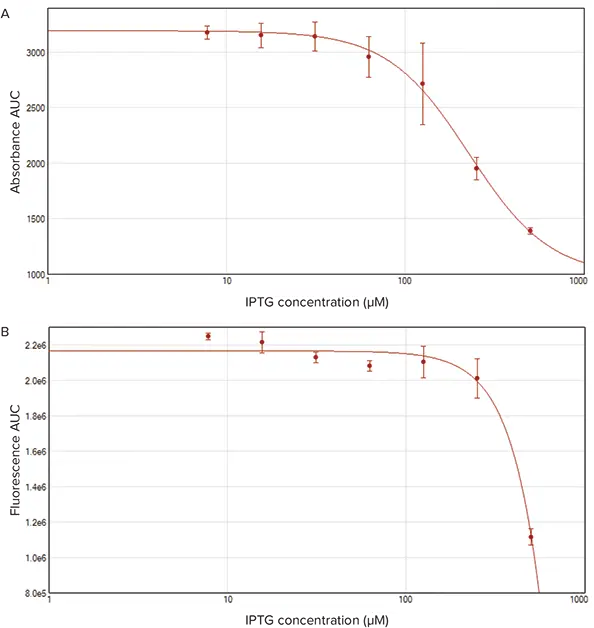
図4. 吸光度と蛍光測定の曲線下面積(AUC)。吸光度(A)と蛍光(B)のAUCをSoftMax Pro 7ソフトウェアで算出し、プロットしました IPTGが細胞増殖(吸光度)とタンパク質発現(蛍光)に与える影響を示すため、4パラメータカーブを適用しました。
RFP/OD600値を算出することで、集団密度に対するタンパク質発現を測定しました。SoftMax Pro 7ソフトウェアを使用してRFP/OD600比を計算し(図5)、動態トレースに基づき、250 µM IPTG処理細菌が最も多くのRFPを細胞あたりで発現しました。
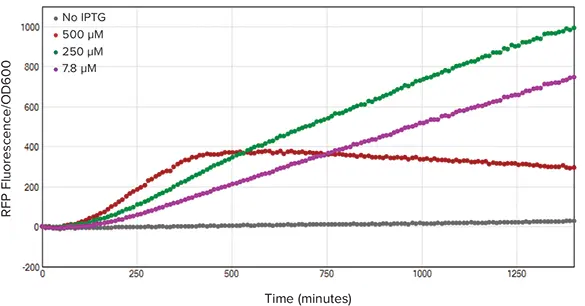
図5. RFP/OD600比。蛍光と細胞密度の比は、蛍光値をOD600値で割ることで算出しました。蛍光を細胞密度で正規化した後、250 µM IPTG処理細菌(緑)が細胞あたりで最も高いRFP濃度を示しました。
結論
本テクニカルノートでは、モレキュラーデバイスのSoftMax Pro 7ソフトウェアとマルチモードマイクロプレートリーダーを用いて、複数の生物学的イベントを時間経過とともに同時に測定できることを示しました。ここで示した実験コンセプトはシンプルですが、より複雑な実験にも応用可能です。
参考文献
- Malakar, P. and Venkatesh, K.V. (2012) Effect of substrate and IPTG concentrations on the burden to growth of Escherichia coli on glycerol due to the expression of Lac proteins. Applied Microbiology and Biotechnology, 93(6), 2543-2549.
- Lee, T.S., Krupa, R.A., Zhang, F., Hajimorad, M., Holtz, W.J., Prasad, N., Lee, S.K., and Keasling, J.D. (2011) BglBrick vectors and datasheets: a synthetic biology platform for gene expression. Journal of Biological Engineering, 5, 12.
PDF版(英語)