Application Note ライブセル蛍光タンパク質測定
非侵襲的イメージング技術
- ライブセルで蛍光タンパク質を容易かつ非侵襲的に測定できます
- 各蛍光色素に最適な結果を得るために波長を調整できます
- 最高の感度を得るためにボトムリードで測定します
PDF版(英語)
はじめに
蛍光タンパク質は、生体内での生物学的イベントをモニタリングするツールとして非常に広く利用されるようになっています。クラゲ Aequorea victoria 由来の緑色蛍光タンパク質(GFP)が最初に選ばれたタンパク質でしたが、現在では、改良型GFPバリアントや他種クラゲ由来の蛍光タンパク質、サンゴ由来のタンパク質など、数多くの選択肢があります。これらは、細菌や酵母から植物、哺乳類まで、さまざまな細胞や生物にクローニングすることができます。蛍光タンパク質は安定性が高く、毒性が最小限であり、外部補因子を必要とせずに生体内で可視蛍光を発する能力を持っています。これらは分子タグや独立したレポーターとして使用でき、タンパク質合成やターンオーバー、タンパク質の転送、遺伝子誘導、細胞系譜など、さまざまな細胞プロセスを可視化、追跡、定量化することが可能です。蛍光タンパク質にはさまざまな色があるため、マルチプレックスアッセイにも利用できます。蛍光顕微鏡やフローサイトメトリーでモニタリングできますが、細胞を物理的にソートしたり細胞内移動を追跡する必要がない場合、マイクロプレート蛍光測定は、より便利でハイスループットな検出システムとして有用です。ここでは、モレキュラーデバイスのマイクロプレートリーダーが、ライブセルにおける蛍光タンパク質を容易かつ非侵襲的に測定できることを示します。
Clontechから3種類のHEK-293細胞株を取得し、それぞれ異なる蛍光タンパク質で安定的にトランスフェクトしました。本研究の目的は、1)3種類の細胞株に対する最適な波長設定を決定すること、2)各細胞株の希釈系列を作成して検出下限(LLD)を推定すること、3)ある細胞株が他の細胞株の存在下で識別可能であることを実証することです。
材料
- 蛍光タンパク質を安定に発現するHEK-293細胞株はClontech社から入手:
◦AcGFP- Aequorea coerelescens(A.victoriaとは異なるクラゲ)からクローニングされたGFPの変異体。
◦ZsGreen-GFPに似ているが、より明るい(サンゴ礁由来)
◦DsRed- サンゴ礁からクローニングされた赤色シフト蛍光タンパク質。 - HEK-293細胞株(非トランスフェクション) (ATCC)
- DME:高グルコース(Irvine Scientific)
- G418: Geneticin (Gibco)
- FBS (Irvine Scientific)
- グルタミン/ペン/Strep溶液 (Gibco)
- トリプシン/EDTA 1X in HBSS (Irvine Scientific)
- 10X ハンク平衡塩溶液 (Gibco)
- 1M HEPES(Irvine Scientific)
- HBSS Buffer(1X Hank's Balanced Salt Solution with 20 mM HEPES buffer): 10X Hank's Balanced Salt Solution(100 mL)、1M HEPES(20 mL)および細胞培養用水(880 mL)から調製。
- 細胞培養用水(Irvine Scientific)
- 黒壁透明底96ウェルマイクロプレート(Corning)または黒壁透明底384ウェルマイクロプレート(Corning)
- SpectraMax® M5 マルチモードマイクロプレートリーダー(モレキュラーデバイス)
- Gemini EMマイクロプレートリーダー(モレキュラーデバイス)
方法
細胞調製と解析
細胞は\(\text{DME} + 10\%\,\text{FBS} + 1\%\,\text{Pen/Strep/L-glutamine} + \frac{500\ \mathrm{µg}}{\mathrm{mL}}\ \text{of G418}\)を含むバルクフラスコで培養しました。非トランスフェクトHEK細胞をコントロールとして含めました。実験前夜にトリプシン処理し、細胞懸濁液を連続希釈して、細胞密度が500,000から100 cells/mLまでになるようにしました。これらを96ウェル(100 µL/well)および384ウェルマイクロプレート(25 µL/well)に一晩播種しました。したがって、播種細胞密度は96ウェルプレートで50,000~10 cells/well、384ウェルプレートで12,500~2.5 cells/wellでした。両方のプレートで各希釈につき12ウェルのリプリケートを設けました。翌日、SpectraMax M5およびGemini EMリーダーでボトムリードとトップリードで測定しました。
波長最適化
SpectraMax M5およびGemini EMはいずれもモノクロメーター搭載のマイクロプレートリーダーです。これらの装置では、各蛍光色素に対して最適なシグナル/バックグラウンド比を得るために励起波長と蛍光波長を調整でき、フィルターベースの装置に比べて優位性があります。一般的な戦略として、励起波長を予想されるEx λₘₐₓより20~25 nm低く設定して蛍光スキャンを行い、蛍光波長を予想されるEm λₘₐₓより20~25 nm高く設定して励起スキャンを行います。この2つのスキャンにより、実際のExおよびEm λₘₐₓ値が得られます。その後、定量解析に最適なシグナル/バックグラウンド比を与える励起波長と蛍光波長の組み合わせを決定するために追加スキャンを行います(ストークスシフトに応じて、励起波長を低く、蛍光波長を高く設定し、不要な励起光を遮断するために蛍光カットオフフィルターを使用します)。場合によっては、2種類のカットオフフィルターの選択のために最終蛍光スキャンを2回行う必要があります。
結果
波長最適化
Gemini EMリーダーでのDsRed波長スキャンを、波長最適化手順の例として以下に示します。励起λₘₐₓを決定するため、蛍光波長を600 nmに設定して励起スキャンを行いました。その結果、励起λₘₐₓは556 nmであることが示されました(図1)。
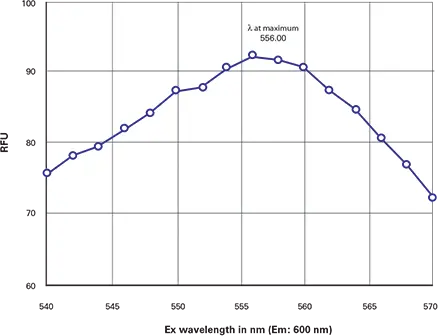
図1. DsRed励起スキャン DsRedの励起スキャンで、λₘₐₓ = 556 nmを示しています。
蛍光λₘₐₓを決定するため、励起波長を535 nmに設定して蛍光スキャンを行ったところ、蛍光λₘₐₓは584 nmであることが判明しました(図2)。ExおよびEmのλₘₐₓ値は、公開されている557/579の値に近いものでした。
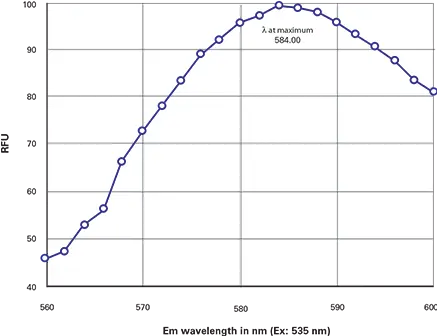
図2. DsRed蛍光スキャン DsRedの蛍光スキャンで、λₘₐₓ = 584 nmを示しています。
次のステップでは、最適なシグナル/バックグラウンド比を得るためのExおよびEm設定を決定しました。ストークスシフトが小さい(22 nm)ため、励起波長を下げ、蛍光波長を上げて両者の分離を広げ、さらに最高感度を得るために蛍光カットオフフィルターを使用する必要があることは明らかでした。そこで、励起波長を550 nmに下げ、570 nmの蛍光カットオフフィルターを選択して、570 nm以上の不要な励起光を遮断しました。575~600 nmの範囲で蛍光スキャンを行ったところ、DsRedトランスフェクト細胞では約587~588 nmにピークが見られました(図3、上の曲線)。バックグラウンド(非トランスフェクト細胞)はその領域で比較的フラットなプロットを示しました(図3、下の曲線)。このスキャンに基づき、最適化された設定はEx/Em = 550/588、575 nm蛍光カットオフフィルターと判断されました。
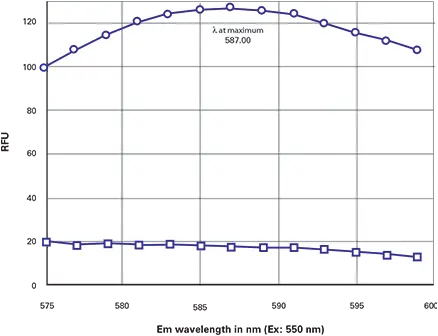
図3. DsRed設定の最適化 蛍光スキャンは570 nmカットオフフィルターを使用し、Exを550 nmに設定して実施しました。上の曲線=トランスフェクト細胞、下の曲線=コントロール細胞。最終的な最適化設定:Ex/Em = 550/588 + 570 nm蛍光カットオフ。
観察されたλₘₐₓ値と、定量解析に推奨される設定を表1にまとめます。
|
Published λ
maxima |
Observed λ
maxima |
M5*に最適化された設定 | |||||
|---|---|---|---|---|---|---|---|
| 蛍光体 |
Ex λ max |
Em λ max |
Ex λ max |
Ex λ max |
Ex | Em | Cutoff |
| AcGFP | 475 | 505 | 480 | 510 | 480 | 525 | 515 |
| ZsGreen | 493 | 505 | 494 | 505 | 494 | 525 | 515 |
| DsRed | 557 | 579 | 556 | 584 | 550 | 588 |
570 |
表1. 観察されたλₘₐₓ値と推奨設定の概要 *SpectraMax M5とGemini EMでは光学系に微妙な違いがあります。Gemini EMでは、AcGFPおよびZsGreenに対する推奨設定はEx/Em 480/510、495 nmカットオフフィルターです。
細胞希釈系列
ボトムリードで測定した希釈系列の結果を図4(96ウェル)および図5(384ウェル)に示します。トップリードの結果は図6(96ウェル)および図7(384ウェル)に示します。ZsGreen細胞株(上の曲線)は他の2種類より約3.5倍明るい結果でした。DsRed細胞株はZsGreenより暗いものの、DsRed波長でのバックグラウンドが低いため、検出下限は両者で類似していました(表2)。
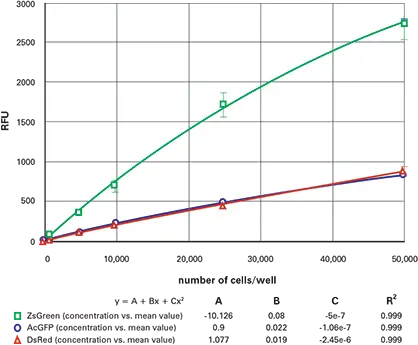
図4. 96ウェル蛍光細胞希釈系列(ボトムリード)ZsGreen(緑)、AcGFP(青)、DsRed(赤)トランスフェクト細胞の96ウェルプレートにおける希釈系列をボトムリードで測定。
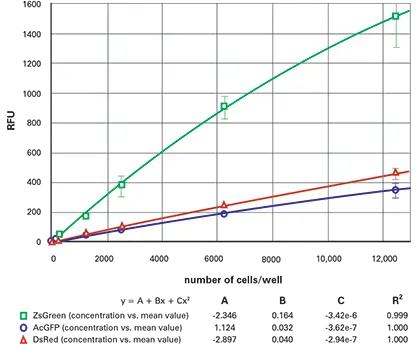
図5. 384ウェル蛍光細胞希釈系列(ボトムリード)ZsGreen(緑)、AcGFP(青)、DsRed(赤)トランスフェクト細胞の384ウェルプレートにおける希釈系列をボトムリードで測定。
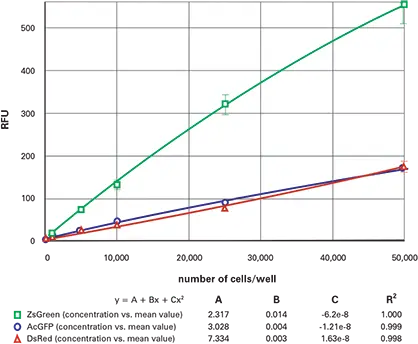
図6. 96ウェル蛍光細胞希釈系列(トップリード)ZsGreen(緑)、AcGFP(青)、DsRed(赤)トランスフェクト細胞の96ウェルプレートにおける希釈系列をトップリードで測定。
| タンパク質 |
ボトムリードを行う(#cells/well)
96-Well 384-Well |
トップリード(#cells/well)
96-Well 384-Well
|
||
|---|---|---|---|---|
| SpectraMax M5 | ||||
| AcGFP | 400 | 250 | 1200 | 600 |
| ZsGreen | 110 | 50 | 350 | 150 |
| DsRed | 200 | 30 | 2000 | 500 |
| Gemini EM | ||||
| AcGFP | 500 | 300 | 1300 | 620 |
| ZsGreen | 110 | 65 | 720 | 170 |
| DsRed | 110 | 30 | 4200 | 200 |
表2. 推定検出下限の概要 *結果は#cells/wellで表記。#cells/mm²に換算するには、96ウェル値を129 mm²/wellで割り、384ウェル値を25 mm²/wellで割ります。
全体的に、プロットはわずかに非線形で、上端で低下しました。これは、高密度の細胞がマイクロプレートの壁に移動し、ビーム外に出る傾向によるものと推測されます。
表2には、各細胞株の推定検出下限(LLD)を示します。LLDの計算式は次のとおりです: \(\text{LLD} = \frac{3 \times \text{SD}_{\text{Blank}}}{\text{slope}}\)。 ここで、\(\text{SD}_{\text{Blank}}\) はブランクウェル(細胞なし培地)の標準偏差、\(\text{slope}\) は曲線の低濃度側の傾きです。 (96ウェルおよび384ウェルプレートでは、それぞれ \(10{,}000\ \text{cells}/\text{well}\) および \(2{,}500\ \text{cells}/\text{well}\) のデータポイントを使用しました)。 非トランスフェクト細胞を含むウェルのRFU信号と標準偏差は、培地のみを含むウェルと類似していました。
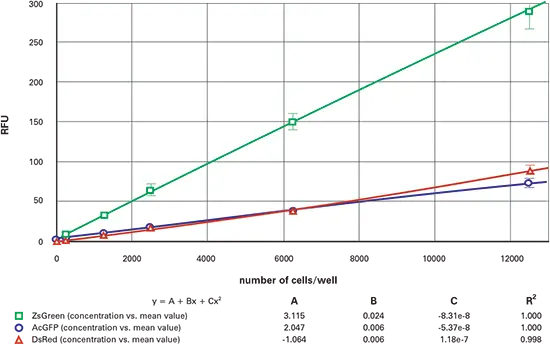
図7. 384ウェル蛍光細胞希釈系列(トップリード)ZsGreen(緑)、AcGFP(青)、DsRed(赤)トランスフェクト細胞の384ウェルプレートにおける希釈系列をトップリードで測定。
この実験では、ZsGreenおよびDsRed細胞株はボトムリードモードで類似した検出下限を示し、両者はAcGFPのLLDより3~4倍低い値でした。この実験は3回実施され、DsRedは常に良好な結果を示したわけではありません。ある実験ではAcGFPと同程度、別の実験ではさらに高いLLDを示しました。これらの差異は、細胞株間で継代回数が異なっていたことに起因すると考えられます(継代を重ねるごとに細胞は暗くなります)。初期の実験ではDsRed細胞株が他よりも早く増殖したため、最初の実験では他の細胞株より2~4回多く継代され、その結果性能が低下したと考えられます。
AcGFPおよびZsGreen細胞株では、トップリードでのLLDは約3倍高くなりました。一方、DsRedではトップリードで10~20倍高くなりました。この感度低下は、DMEMが顕著な赤方偏移蛍光信号を持つことによる干渉と考えられます。実際、着色培地を無色のHBSSに置換すると、DsRedのトップリード結果は改善しました。
表3には、培地を除去し、無色のバッファーに置換した以前の実験結果を示します。ボトムリードの結果は変化しませんでした(ただし、AcGFPのLLDは改善したように見えました)。これは、この操作によって細胞の顕著な損失がなかったことを示しています。一方、トップリードの結果は約3倍改善しました。これらの結果は、トップリードモードで感度が低下する原因の一部が、培地の着色にあるという仮定を支持しています。
| ボトムリード | トップリード | |||
|---|---|---|---|---|
| 蛍光体 | メディア | バッファ | メディア | バッファ |
| AcGFP | 900 | 250 | 16,000 | 3300 |
| ZsGreen | 40 | 70 | 1400 | 400 |
| DsRed | 750 | 600 | 8200 | 3800 |
表3. 培地を無色HBSSに置換した場合の検出下限(#Cells/Well)への影響(Gemini EMリーダーで96ウェルマイクロプレートを使用)
細胞混合実験
この実験では、96ウェルプレートの各ウェルにさまざまな細胞混合物を入れ、各ウェルの総細胞数は50,000細胞としました。したがって、1:1混合では各細胞タイプが25,000細胞、1:1:1混合では各細胞タイプが16,700細胞となります。プレートはEx/Em 480/510(AcGFPおよびZsGreenに最適)および550/588(DsRed設定)で測定しました。観察されたRFU値は予測値と類似していました。結果は、DsRedの存在下でAcGFPまたはZsGreenを測定でき、その逆も可能であることを示しています(表4)。
| Cells | RFU観察値/予測値(%) |
|---|---|
| AcGFPとZsGreenでの結果: 設定 (480/510) | |
| Ac + Ds | 92.8 |
| Zs + Ds | 99.0 |
| Ac + Ds + Zs | 97.8 |
| DsRedでのRFU結果:設定(550/588) | |
| Ac + Ds | 94.9 |
| Zs + Ds | 83.7 |
| Ac + Ds + Zs | 99.6 |
表4. 細胞混合物で得られた結果
結論
モレキュラーデバイスのマイクロプレートリーダーは、接着したライブセルにおける蛍光タンパク質を容易に検出できます。各蛍光色素に最適な結果を得るために波長を調整できる利点があります。ボトムリードはより高い感度を提供しますが、トップリードでも実用的な結果が得られ、特にZsGreenおよびDsRedを含む細胞株で有効です。DsRed細胞株のトップリード結果は、培地を無色のHBSSバッファに置換することで改善できます。
PDF版(英語)