Application Note 患者由来大腸がんオルガノイドによる
ハイスループット創薬支援技術
- アッセイ対応済みの患者由来オルガノイドを使用することで、オルガノイドモデルの培養時間を短縮
- ハイコンテントイメージングシステムを用いて、化合物曝露に伴うオルガノイドの形態変化を経時的に検出
- 形態学的なリードアウトに対応したディープラーニングツールによる堅牢な自動イメージ解析パイプラインを構築
PDF版(英語)
はじめに
Angeline Lim, Jason Baade, Aditya Katiyar, Prathyushakrishna Macha, Zhisong Tong, Oksana Sirenko|モレキュラーデバイス
Elizabeth Fraser | Cellesce社
患者由来オルガノイド(PDO)は、創薬パイプラインにおける脱落率を低減する有望なツールとして注目されています。これらの腫瘍オルガノイドは、3D腫瘍の多細胞ミニレプリカであり、in vivoの特性を保持していることが示されています *1。研究によれば、患者とその由来オルガノイドは薬剤に対して類似した反応を示すことが確認されており、PDOは治療効果および毒性の予測において、生物学的に関連性の高い高度なin vitroモデルとされています。しかし、アッセイの再現性、スケーラビリティ、コストといった課題により、PDOの主流創薬パイプラインへの導入は限定的でした。
このスケーラビリティの課題に対応するため、Cellesce社はPDOの大規模増殖を可能にする独自の産業用バイオプロセスを開発しました。オルガノイドは、栄養素および成長因子が継続的に供給され、培養中に毒素が蓄積して細胞死を引き起こすことを防ぐ環境下で培養されます。この独自技術により、高度に標準化されたアッセイ対応済みPDOをスケールで生成することが可能となり、創薬のためのハイスループットスクリーニングなどの応用が実現します。
これらのPDOのハイスループットアプリケーションにおける有用性を示すため、大腸がん(CRC)PDOをマニュアル(384ウェルプレート)および自動化(96ウェルプレート)手法でマルチウェルプレートに播種しました。自動化には、BAB400バイオプリンターのピペッティング機能を使用し、オルガノイドを混合したMatrigelドームを各ウェルの中央に分注しました。オルガノイドには、選択された抗がん剤を様々な濃度で処理しました。PDOは透過光イメージングにより経時的にモニタリングされました。解析には、ディープラーニングベースの画像セグメンテーションモデルを開発・使用し、サイズ、テクスチャ、強度などの形態学的および表現型的リードアウトを取得しました。さらに、ライブ/デッド細胞染色を用いた生存率アッセイを実施し、ハイコンテント共焦点イメージングでPDOを3Dイメージングしました。
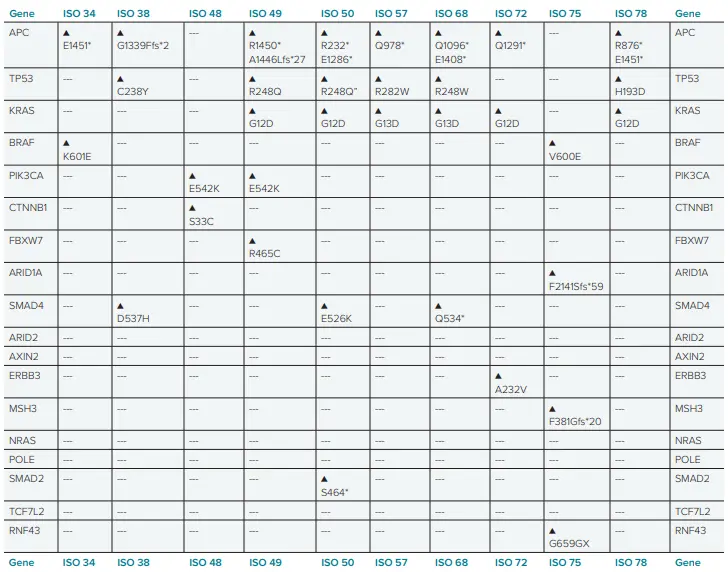
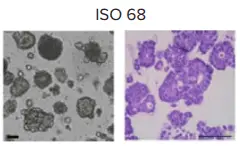
図1.患者由来の大腸がんオルガノイド(Cellesce)は、凍結保存バイアル(100,000オルガノイド)で提供されます。各ラインには、変異プロファイル、腫瘍特性、形態情報などのデータが付属しています。
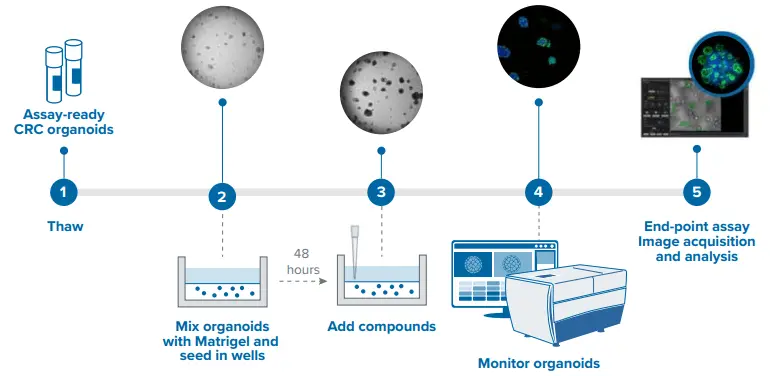
図2.アッセイ対応済み大腸がんオルガノイドの使用ワークフロー
材料と方法
細胞培養
大腸がんオルガノイド(ISO68ライン、Cellesce)は、メーカーの指示に従って取り扱いました(図1)。概要としては、オルガノイドを37°Cで迅速に解凍し、培地で穏やかに懸濁・洗浄しました。オルガノイドを含むペレットをMatrigelに再懸濁し、384ウェルプレートに1ウェルあたり200オルガノイドで播種しました。回復を促進するため、ROCK阻害剤を含む培地で48時間インキュベートしました。その後、選択した化合物を様々な濃度で5日間、4重測定で処理しました(図2)。
自動播種には、BAB400(Advanced Solution)のピペットツールを用いて、オルガノイドを混合したMatrigel懸濁液を96ウェルプレートに播種しました。グリッパー(PnPツール)シーケンスを使用してプレートの蓋の開閉や、プレートをImageXpress® Micro Confocal ハイコンテントイメージングシステムへ移送する操作を行いました。各ウェルの中央に7 µLのオルガノイド懸濁液を分注し、「ドーム」を形成しました。自動化のためのツール座標のマッピングは、実行前に設定・記録されました。
ハイスループットイメージングと解析
化合物処理の効果は、ImageXpress® Micro Confocalシステムを用いて経時的にモニタリングされました。CRCオルガノイドは4X対物レンズでzスタックを有効にしてイメージングされました。生存率アッセイでは、Hoechst、Calcein AM、エチジウムホモジマーを用いて、37°Cで2時間インキュベートしました。画像は10X対物レンズでzスタック付きで取得しました。ファロイジン染色では、オルガノイドを4% PFAで固定後、phalloidin488で染色し、10Xまたは20X(水浸対物レンズ)でイメージングしました。
画像解析にはIN Carta® 画像解析ソフトウェアを使用し、ディープラーニングベースの手法でオルガノイドのセグメンテーションモデルを構築しました。
結果
大腸がんオルガノイドによるアッセイ構築
アッセイ対応済みがんオルガノイドの利用を評価するため、384マイクロウェルプレートを用いた概念実証スタディを設計しました。オルガノイドを解凍後、Matrigelと混合して播種しました。48時間後、CRCオルガノイドに対して9種類の化合物を7段階の濃度で、4重測定にて処理しました(図3)。5日後、生存率アッセイを実施し、各化合物がオルガノイドの生存率に与える影響を評価しました(図5)。生存率アッセイの後、オルガノイドを固定し、ファロイジン染色を行って、化合物がオルガノイドの形態に与える影響を観察しました(図6)。
図3. アッセイのセットアップ 384ウェルプレートに播種されたオルガノイドのプレートマップビューを示しています。挿入図は、1つのウェルから取得された代表的な画像で、上段が蛍光画像、下段が透過光画像です。オルガノイドは、Hoechst(核、青)、ファロイジン488(アクチン、緑)、エチジウムホモジマー(死細胞マーカー、赤)で染色されています。
ディープラーニングを用いたオルガノイドの表現型変化のモニタリング
PDO(患者由来オルガノイド)は蛍光マーカーを発現していないため、成長の経時的な観察には明視野イメージングを用いました(図4)。オルガノイドの品質を評価するために、取得した画像をディープラーニングベースのセグメンテーション手法で解析しました。CRCオルガノイドの成長は、直径や面積の変化を測定することでモニタリングできます。また、化合物の影響も定量化可能です。本研究では、ロミデプシンおよびトラメチニブによってCRCオルガノイドの成長が抑制されました。
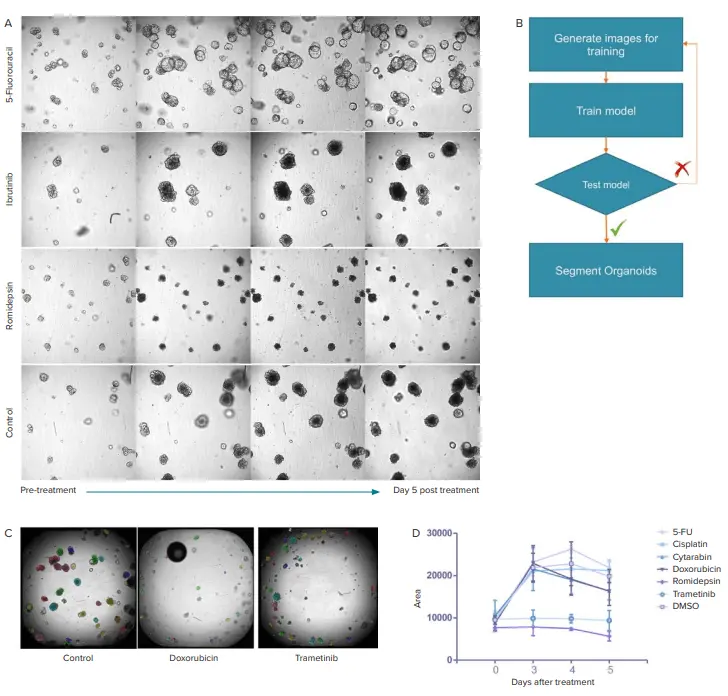
図4. A)Matrigel中のCRCオルガノイドの代表的な画像。化合物処理後、5日間にわたり観察を行いました。B)IN Cartaソフトウェア内のSINAPワークフローの概要。オルガノイドのセグメンテーションモデルを生成するためのプロセスを示しています。C)透過光画像は、非均一な背景やエッジ効果、気泡などのアーティファクトが多く、堅牢なセグメンテーションを妨げます。ここでは、セグメンテーションマスクを重ねた代表的な画像を示しています。SINAPモデルによりCRCオルガノイドのセグメンテーションを実現しました。D)DMSOコントロールと各化合物(5-FU 5µM、シスプラチン 2.5µM、シタラビン 50µM、ドキソルビシン 15µM、ロミデプシン 2.5µM、トラメチニブ 5µM)処理群における、5日間の平均オルガノイド面積の推移を示すグラフ。Day 0は化合物処理前の画像取得時点を示します。エラーバーはウェル間の標準偏差を表します。
化合物効果の定量化のための生存率アッセイ
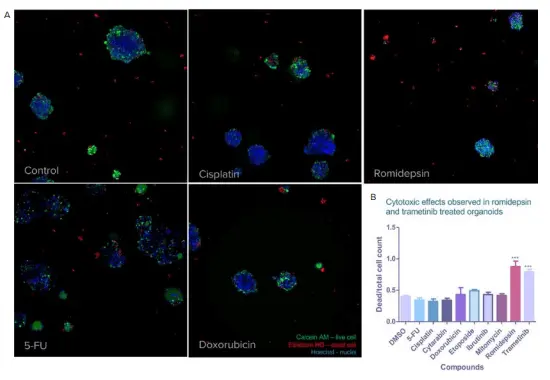
図5. A)化合物処理後のCRCオルガノイドに対して生存率アッセイを実施し、染色およびイメージングを行いました。Calcein AM(生細胞、緑)、エチジウムホモジマー(死細胞、赤)、Hoechst(全核、青)で染色された代表的な画像を示しています。B)各化合物処理群における死細胞(Hoechstおよびエチジウムホモジマー陽性)の割合を示しています。ロミデプシンおよびトラメチニブ処理群では、コントロールと比較して有意に死細胞が増加しました(p<0.001)。
CRCオルガノイドにおける化合物誘導性の表現型変化
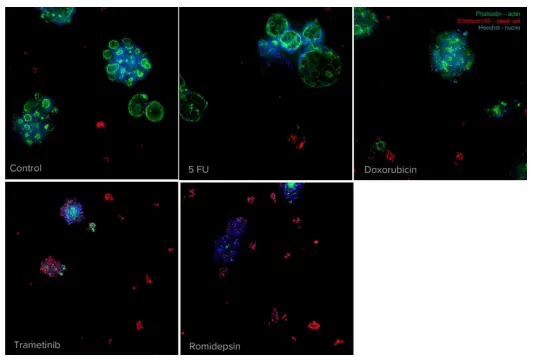
図6.化合物処理後5日目にオルガノイドを固定し、ファロイジンで染色して表現型の変化を評価しました。
CRCオルガノイド播種の自動化
ワークフローの自動化の可能性を検討するため、BAB400バイオプリンターを用いて96ウェルプレートへのCRCオルガノイド播種を最適化しました。Matrigelと混合したオルガノイドを、ピペットツールを用いて各ウェルの中央にドーム状に播種しました(図7)。
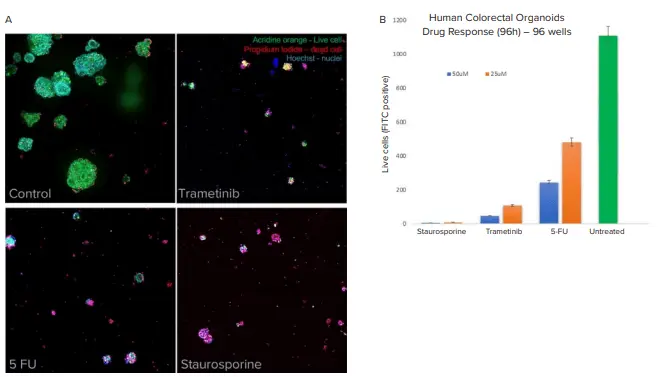
図7. A)Day 2にトラメチニブ、フルオロウラシル(5-FU)、スタウロスポリン(各50µMおよび25µM)で処理し、Day 6に染色およびイメージングを実施しました。B)薬剤処理後のオルガノイド1個あたりの平均生細胞数を定量化しました。
結論
- 本研究の結果は、PDOを化合物スクリーニングのようなハイスループットアッセイに活用できることを示しています。標準化されたアッセイ対応PDOの利用により、3Dモデルを用いたスクリーニングにおいて大幅な時間短縮が可能となります。
- また、AIベースのアプローチにより、ラベルフリーの生物学的モデル(例:3Dオルガノイド)に対しても堅牢なセグメンテーション解析が可能であることを示しました。
参考文献
- Vlachogiannis G, Hedayat S, Vatsiou A, et al. Patient-derived organoids model treatment response of metastatic gastrointestinal cancers. Science. 2018;359(6378):920-926. doi:10.1126/science.aao2774
PDF版(英語)